DNA测序技术,又叫基因测序技术。测序技术是基因组学研究的基础和核心技术,是对DNA分子的核苷酸排列顺序的测定。
(一)第一代测序技术
双脱氧链终止法(the chain termination method)由英国剑桥分子生物学实验室的生物化学家桑格等人于1977年发明,该技术的发明标志着第一代测序技术的诞生。除了双脱氧链终止法,第一代测序技术还包括开启了大片段DNA序列快速测定先河的化学降解法。
第一代测序法的优势是其准确率和读长,适合对新物种进行基因组长距离的框架构建及测序长度为kb~Mb级别的小规模项目,在人类基因组计划的后期发挥了关键作用,第一代测序技术参与并完成的划时代的研究成果见表9-1。
表9-1 第一代测序技术参与的研究成果

1.Sanger测序法 Sanger测序法,又称双脱氧链终止法或酶法,是一种基于DNA聚合酶合成反应的测序技术。
(1)基本原理:生物体内,在DNA聚合酶作用下,以单链DNA为模板,以寡聚核苷酸为引物并将测序引物一端进行放射性标记,将脱氧核糖核苷酸(dNTP)底物加到引物的3′-OH端,使引物延伸链的延长通过引物的3′-0H基,与脱氧核糖核苷酸底物的5′-磷酸基团,生成磷酸二酯键,通过这种磷酸二酯键的不断形成,新的互补DNA得以从5′→3′延伸。在体系中加入2,3-双脱氧的A,C,G,T核苷三磷酸,即称为ddATP,ddCTP,ddGTP,ddTTP的底物时,该底物的5′-磷酸基团是正常的,能够加到正常核苷酸的3′-OH基末端,但其自身3′-OH基团由于脱氧而不存在,下一个核苷酸不能通过5′-磷酸与之形成磷酸二酯键。在4组独立的酶反应体系中,在底物dNTP中分别加入4种ddNTP中的一种,在掺入ddNTP的位置链延伸终止。结果会产生4组分别终止于模板链的每一个A、每一个C、每个G和每一个T位置上的具有相同5′-引物端和以ddNMP残基为3′端结尾的一系列长短不一的混合物。通过高分辨率变性聚丙烯酰胺凝胶电泳,区分长度差一个核苷酸的单链DNA,从放射自显影胶片上可直接读出DNA上的核苷酸顺序。
(2)双脱氧链终止法的反应体系:
1)模板:纯化的单链DNA或热变性、碱变性的双链DNA,注意要保证纯度和浓度,特别是双链DNA模板。
2)引物:人工合成的寡核苷酸,长度为18-22bp。设计引物时需尽量避免3个以上相同碱基重复,特别是G或C,Tm值范围为55~60℃,减少发夹结构和引物二聚体形成。
3)DNA聚合酶:选用合适的DNA聚合酶对保证测序质量非常重要。常用的几种酶:①大肠杆菌DNA聚合酶Ⅰ大片段(Klenow片段),此酶是最早用于建立桑格测序的酶。②测序酶(sequenase),该酶是一种经过化学修饰的T7噬菌体DNA聚合酶,其原来具有很强的3′→5′外切酶活性,但经化学修饰后大部分被消除。③耐热DNA聚合酶,耐热DNA聚合酶应用于以桑格双脱氧链终止法,聚合酶链式反应PCR常用该酶。
4)放射性标记dNTP:传统的DNA测序方法都采用α-32P-dNTP作为放射性标记物,近年来α-35S-dNTP被广泛采用,是由于35S产生较弱的β射线,放射自显影图谱具有较高的分辨率和较低的本底,测序反应产物可在-20℃保存一周,而分辨率并不下降。
5)ddNTP:32P标记或35S标记的四种ddNTP。
6)用于测序的变性凝胶电泳:①胶长40cm;②厚度均匀;③浓度为4~8%聚丙烯酰胺凝胶;④7mol/L尿素。
(3)双脱氧链终止法的操作流程:
①利用基因工程的方法获取一定数量的单链待测DNA模板。
②在待测DNA片段的3′端合成一段互补的寡核苷酸引物。
③制胶。
④将待测序列的DNA模板、引物、DNA聚合酶、4种dNTP、一种带放射性同位素标记的脱氧核糖核酸(如α-32P-dATP)混合。将此反应物分为4等份,每份内再加入一定比例的一种ddNTP,如此形成A、T、C、G四个反应体系,最后在各反应体系中加入DNA聚合酶催化DNA片段合成。
⑤模板引物退火,引物与DNA单链模板的3′端进行配对。
⑥在适当温度条件下延伸互补链,就可得到四组分别以A、G、C、T、终止的长短不一的互补链的混合物。
⑦将制得的四组混合物全部平行地点样于变性聚丙烯酰胺凝胶电泳板上进行电泳,每组一泳道,四条泳道相邻并列,每条泳道上按产物分子量小→大从正极到负极(胶板的下→上)分离开来,依次排列。由于各产物均带有放射性标记,以上结果经放射自显影,从而制得相应的放射性自显影图谱。
⑧将电泳凝胶与X光胶片压在一起,于低温下自显影数天,即可得到放射自显图谱,通过该图谱即可直接读出核苷酸的排列顺序。
(4)大片段DNA的操作策略:
1)随机法(又称鸟枪法):将长链DNA随机断裂成适于测序的片段,主要方法:①超声波处理;②DNase I切割;③限制酶消化。随后将随机切割出的子片段分别插入到载体的多克隆部位,进行克隆扩增,对不同的片段分别测序,然后将它们重叠排列,便可连出整体DNA的全长序列,或将单个片段序列输入计算机处理,可整合出整个DNA序列。
2)嵌套缺失法(又称互套缺失法):该法的基本原理是基因DNA链的一端与载体相连固定,另一端在核酸外切酶的作用下随着时间的延长,较匀速地消化变短,这样可人为获得一组长度不等的从一端开始缺失的DNA片段。然后从缺失端开始测序,这样每段的基因起始部分的序列总是与前一基因片段序列的后面部分重合,这样彼此套接可以很准确地测出整个基因的DNA序列。
3)引物延伸法:该法是一种定向测序法,基本原理是从基因的3′端,依赖特定引物可合成一定长度的互补DNA链,用于测序。再根据该序列,设计新的寡核苷酸作为下一延伸段的引物,如此逐步向前推进,最终测得目的基因的全长序列。
2.化学降解法 与Sanger法不同,化学降解法不需要进行酶催化反应,不会由于酶催化反应而带来误差,能够对未经克隆的DNA片段直接测序,特别适用于测定G和C含量较高的DNA片段,以及短链的寡核苷酸片段的序列。
(1)基本原理:首先需要对DNA进行化学降解,在进行测序前由双链变为单链,对待测DNA片段的一端进行放射性标记,然后将标记DNA分为G、A+G、C+T及C,共4组反应体系,用与碱基发生专一性反应的化学试剂处理反应体系,即不同的化学试剂处理不同反应体系,通过相互独立的化学反应,分别特异性地针对某一种或某一类碱基进行切割,每组中的每个片段都有放射性标记的共同起点,但长度取决于该组反应针对的碱基在原样品DNA分子上的位置,从而产生一系列长度不同且一端被标记的DNA片段,然后各组反应物通过凝胶电泳进行分离,通过放射自显影检测末端标记的分子,确定各片段末端碱基,并直接读取待测DNA片段的核苷酸序列。
(2)化学法测序的基本步骤:①限制酶消化待测DNA得到长度为100-200bp的一组片段,纯化回收每1个片段作为测序材料。②碱性磷酸酶处理消除5′磷酸。③多核苷酸酶催32PdNTP标记(5′-OH)末端。④使标记片段变性为单链。⑤分成4-5个反应体系分别进行化学处理,严格控制反应条件。DNA链上每50-100个碱基只有1个被裂解,这样,每个反应中虽然都在同一核苷酸处断裂DNA链,但由于碱基位置不同,产生1组不同长度的DNA片段。其长度可由几个核苷酸到接近待测DNA全长。⑥聚丙烯酰胺凝胶电泳。⑦放射自显影。⑧4个反应管统一阅读,DNA 4个碱基每个位置都有一个相应的片段,待测DNA全部核苷酸序列就可直接读出。
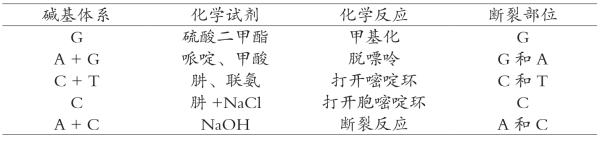
(3)化学测序法的特异切割:在化学降解法中,专门用来对核苷酸作化学修饰并打开碱基环的化学试剂主要有肼(hydrazine)、硫酸二甲酯(dimethylsμLphate)、哌啶、甲酸、联氨等。表9-2列出了化学降解法常用化学试剂。
表9-2 化学降解法测序的常用化学试剂
(二)第二代测序技术
随着人类基因组计划的完成,基于第一代测序方法在原始数据质量和序列读长方面的优势,不会很快消失,将与新的测序方法并存,但由于速度慢、通量低、成本高等原因,已经无法满足大规模基因组测序高自动化、高通量、低成本的要求,这就促使了以高通量为显著特点的第二代测序技术——超大规模多模板平行测序技术的诞生,可以一次就完成几十万到几百万条DNA分子的测序,使得基因组深度测序和转录组测序变得简单了。第二代测序技术的核心思想是边合成边测序(Sequencing by Synthesis),即通过捕捉新合成的末端的标记来确定DNA的序列,现有的技术平台主要包括Roche/454 FLX、Illumina/Solexa Genome Analyzer和Applied Biosystems SOLID system。
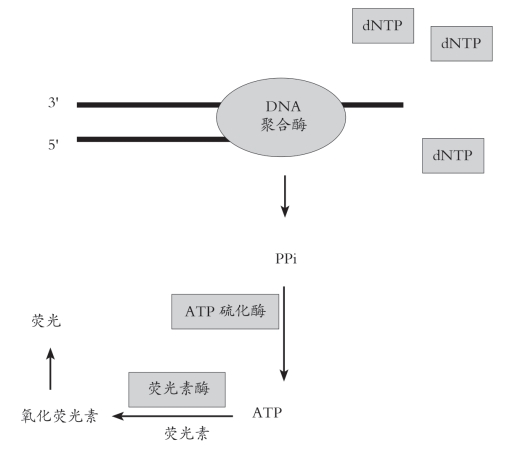
1.焦磷酸测序 焦磷酸测序(Pyrosequencing)技术于1987年由454公司发明,它是新一代DNA序列分析技术,是首个实现商业化的第二代测序技术。该技术利用dNTP在DNA聚合反应时释放的焦磷酸基团(PPi)信号进行测序,该技术不需要电泳,也不需要荧光标记,因此无须荧光分子的激发和检测装置。该方法在油溶液包裹的水滴中扩增DNA,即emulsion PCR。开始时,每一个水滴中仅包含一个包被大量引物的磁珠和一个连接到微珠上的DNA模板分子。将扩增的DNA产物加载到特制的PTP板上,板上有上百万个孔,每个微孔只能容纳一个磁珠。在测序时,向反应体系中循环加入一种dNTP,如果该dNTP与待测模板配对,在DNA聚合酶的作用下,dNTP插入到延伸的DNA链时,释放出一个PPi(焦磷酸分子);在ATP硫酸化酶催化下,PPi与5′-磷酸硫酸(APS)生成一个ATP分子;ATP分子在荧光素酶(Luciferase)的作用下,将荧光素(luciferin)氧化成氧化荧光素(oxy luciferin),同时产生的可见光信号,被CCD光学系统捕获,获得一个特异的检测信号,信号强度与相应的碱基数目成正比。通过按顺序分别并循环添加四种dNTP,读取信号强度和发生时间,实现DNA序列测定。剩余的dNTP和ATP由三磷酸腺苷双磷酸酶降解。总反应体系包括:测序引物、DNA模板、酶混合物(DNA聚合酶、ATP硫酸化酶、荧光素酶、三磷酸腺苷双磷酸酶)和荧光素。上述原理参见图9-1所示。
 (https://www.daowen.com)
(https://www.daowen.com)
图9-1 焦磷酸测序原理图
焦磷酸测序技术可以快速、准确地进行短DNA序列分析,便于构建标准化操作流程,很适合大样本的快速检测,省时省力、费用低,目前已经逐渐成为实验室非常重要的测序手段,受到越来越多的关注,在遗传学分析、SNP分析、分子诊断、细菌与病毒分型、药物基因组学等方面都有广泛的用途。但焦磷酸测序技术在原理上存在着缺陷,测序过程中没有终止基团可以停止DNA链的延伸,在测定一连串相同碱基区域时,如一连串的AAAA……,仅依靠光信号的强度来推断其长度,会出现少读或多读一个至几个碱基的情况。
2.可逆末端终止法——改进的“边合成边测序技术” 在Sanger的双脱氧末端终止法测序方法的基础上,经过改进而发展起来的可逆末端终止法(Sequencing by Reversible Terminator),已被用于测序平台Illumina Genome Analyzer中。基本原理是用不同的荧光物质分别对四种dNTP进行标记,这样每种dNTP就会释放出不同的荧光信号,当DNA聚合酶合成互补链时,每次测序反应只有单个碱基被延伸,将捕捉的荧光信号经过专门的处理软件处理。每步反应完成后,用特异的酶将荧光标记物除去,便可进行下一个碱基的测序,这样就可获得待测DNA的序列信息。
该方法的最大优点在于将读长和读取数据的灵活性有机结合起来,具有准确度高、通量高、成本低等优点,并且可以对DNA分子进行双末端测序。主要的缺点是由于光信号衰减和移相的原因使得序列读长较短,而且测序中每一步化学反应都可能失败,例如:不能将荧光标记物除去而导致一些DNA链过长而另一些DNA链没有同步延伸,进而引起信号衰减或荧光信号移相,而且错误率是累积的,即DNA链越长错误率越高,这些都限制了读长的增加。
目前,经过更新,此测序方法的读长由开始的35个碱基提高到100多个碱基,通量也达到50G。Illmina在2010年推出了HiSeq2000,使测序成本大大降低,通量达到200G,且有进一步提高的潜力。
3.连接测序 2005年,Church实验室提出了连接测序法(Sequencing by Ligation,SBL),利用DNA连接酶在连接过程中进行测序,以四色荧光标记寡核苷酸进行连续的连接反应为基础。随后,ABI公司将该技术发展成商业化的测序技术——SOLiD(Sequencing by Oligonucleotide Ligation and Detection)。
基本原理是以DNA连接酶替代DNA聚合酶,用部分简并了的寡聚核苷酸替代dNTP。首先,把待测序列打断成很小的DNA片段,在小片段两端加上不同的接头,连接载体,构建ssDNA文库;然后ssDNA进行PCR扩增,并对扩增产物进行3′端修饰,在反应体系中加入连接酶和通用测序引物进行测序,由于每轮反应仅能确定特定位置的碱基,每轮反应结束后测序引物必须向前移动一个碱基,如此循环直到读完所有序列。
连接测序法为两碱基测序(two base encoding),每个位置均被重复扫描两次,由于采用连接酶,保真度和准确度较高,准确度可达99.94%,每天最多数据产出量可达20G。
4.杂交测序技术 杂交测序法(Sequencing by Hybridization,SBH)是在20世纪80年代末90年代初出现的一种新的测序方法,利用DNA芯片技术,将一系列已知序列的单链寡核苷酸片段固定在基片上,把待测的DNA样品片段变性后与其杂交,根据产生的杂交图谱排列出样品的序列。
SBH概念的提出是发展DNA芯片技术的初衷,其过程是将寡核苷酸探针固定到芯片上,待测样品中带有标记的DNA靶标与之配对,当配对的DNA有至少1个碱基的差异时,就会影响到杂交时的荧光信号强度,根据荧光信号便可知待测的DNA序列上相应的碱基特征。在实际操作中,杂交必须保证能够区分完全匹配和不完全匹配的探针,理论上,完全匹配探针的杂交信号比含错配DNA的杂交信号要强。
SBH具有快速、成本低、易于自动化等优点,但最佳杂交条件很难统一,易出现假阳性和假阴性的杂交结果,不能准确地排列重叠序列,所以,该方法不适用于含有许多内部重复和简单序列单元的DNA。目前该方法主要用于再测序,如SNP检测、突变分析、疾病分子诊断、基因分型等。
(三)第三代测序技术
第二代测序技术极大地推动了基因组学的发展,广泛地应用于各研究领域,但它是通过读取碱基连接到DNA链上时释放的光学信号间接确定的,对仪器设备要求比较高,且试剂、耗材在测序成本中的比例相当大,读长较短,对后续的序列拼接、组装以及注释等生物信息学分析带来困难。另外,第二代测序技术以PCR为基础,扩增后DNA片段的数目和扩增前DNA片段的数目会有偏差,这对基因表达分析有很大的影响,对大量表达的基因的影响会更大。在此情况下,第三代测序技术应运而生,数据读取速度更快,读长更长,不需要PCR扩增,测序成本更低。第三代测序技术的标志是单分子测序和纳米孔技术。
1.单分子测序 2008年,美国科学家Harris发明了单分子测序法(Single MolecμLe Sequencing,SMS),该方法基于边合成边测序的思想,首先对模板DNA进行修饰,测序时,依次加入用不同的荧光素标记的不同的碱基,在DNA聚合酶的作用下,核苷酸配对到相应的模板上,激发出荧光,根据采集到的荧光信号确定DNA序列。单分子测序法突破了建库扩增的限制,可以直接读取DNA序列碱基,这与传统的荧光测序法是不同的,因此测序速度大大提高。
2.单分子实时测序 2009年,Pacific Biosciences(PacBio)公司成功研发单分子实时测序技术(Single-molecμLe Real-Time Sequencing,SMRT),该技术有两大特点:一是单分子水平的边合成边测序,二是实时,保证了测序的高精确度和高速度,只要一条DNA模板,在碱基合成的过程中就可读出序列。这种技术的核心是零级波导技术(Zero-Mode Waveguides,ZMW),测序前每种dNTP都标记不同的荧光物质,且由传统碱基标记改为磷酸基团标记。每个ZMW孔内固定有DNA聚合酶,由于ZMW孔只允许单个核苷酸通过且只有与模板匹配的碱基与聚合酶结合,在荧光检测区被激光束激发出荧光,而其他核苷酸由于停留时间短,不能进入荧光检测区,也就不会干扰DNA序列信息的读取。这就好像在DNA聚合酶上安装了一个电子眼,直接观察每个合成上是何碱基。
SMRT完全依靠 DNA聚合酶,不需要其他化学物质中断聚合反应,是真正的实时测序,荧光基团标记在磷酸基团上,消除了DNA合成过程中的空间位阻,测序速度可达每秒10个碱基,延长了测序读长,理论上可以达到几千个碱基。如果将DNA聚合酶换成RNA反转录酶,还可以实现对RNA的直接测序。
3.纳米孔测序 美国科学家Daniel B.和David D.发明了一种新的测序技术——纳米孔单分子测序技术(Nanopore Sequencing),是近年来发展最快、最热门的技术之一。其测序原理如图9-2,测序时使用的纳米孔每次只允许一个核苷酸通过,将这种纳米级小孔放在电解液中,很容易就可测定通过纳米孔的电流强度,当DNA分子的核苷酸依次通过纳米孔时,通过纳米孔的电流会被阻断而变弱,由于不同核苷酸的空间构象不同,因此阻断电流变弱的程度也不同,检测电流变弱的程度就可判断对应的碱基是什么,从而实现实时测序。这种技术类似电泳,有很好的持续性和准确性,可以在很短的时间内完成基因组测序。
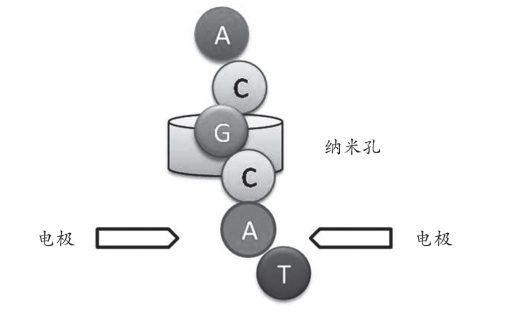
图9-2 纳米孔测序原理图
(四)高通量测序和全基因组测序
近年来,随着科技的发展以及研究的深入,高通量测序技术以及全基因组深度测序技术越来越受到重视,增加了课题研究的深度以及实验结果的可靠性。
1.高通量测序技术 高通量测序技术(High-Throughput Sequencing,HTS)又称为下一代测序技术(Next Generation Sequencing,NGS),以能一次并行对几十万到几百万条DNA分子进行序列测定等为标志,虽然出现时间不长,但发展很快。高通量测序技术是对传统测序的一次革命性的改变,可以轻松实现一次对几十万到几百万条DNA分子进行序列测定,也被称为深度测序(deep sequencing)技术。根据发展历史、影响力、测序原理和技术不同等,主要有以下几种:大规模平行签名测序(Massively Parallel Signature Sequencing,MPSS)、聚合酶克隆(Polony Sequencing)、454焦磷酸测序(454 pyrosequencing)、Illumina(Solexa)sequencing、ABI SOLiD sequencing、离子半导体测序(Ion semiconductor sequencing)、DNA 纳米球测序(DNA nanoball sequencing)等。
高通量测序在基因组学研究中的应用主要集中于两个方面,一方面是基因组从头测序(denovo sequencing),不需要已知的参考DNA序列,可以直接对生物的全基因组序列进行测定,利用生物信息学的方法对序列进行拼接、组装,从而获得该物种的全基因组图谱。另一方面是基因组重测序,是对不同个体进行基因组测序,在此基础上对个体或群体进行差异性分析。随着基因组学在医学研究领域的不断拓展,对致病突变的研究由外显子区域扩大到全基因组范围,在全基因组水平上检测疾病关联的常见、低频,甚至是罕见的突变位点,以及结构变异等,具有重大的科研和产业价值。
2.全基因组测序 全基因组测序(Whole Genome Sequencing)是通过生物信息学手段,运用新一代的高通量DNA测序仪来分析不同机体基因组间的结构差异,并同时完成对单核苷酸多态性(SNP)及基因组结构的注释。
全基因组测序技术的出现对医学领域来说是一次革命性的进步,已经成为疾病研究、临床诊断中重要的手段。通过全基因组测序获得碱基全序列,便于进行全面、精确的分析,破解其包含的信息,加深对疾病发生机制的了解,有针对性地制定相应的应对办法。目前,全基因组测序主要应用于癌症、传染性疾病和遗传性疾病的致病机理的研究方面。通过全基因组测序,可以对癌症中体细胞突变进行鉴定,为癌症的诊断和治疗提供了一种有效的方法,使得癌症的治愈也将成为可能;可以为研究遗传性疾病的发病机制和发现新的突变体提供新的理论依据,对于查找遗传疾病提供新的分子途径,为疾病的预防和治疗提供了新的方向。在疟疾、结核病等传染性流行病学调查和耐药机制的研究中也广泛采用全基因组测序技术,为这些疾病的预防、诊断和治疗提供了新的思路。
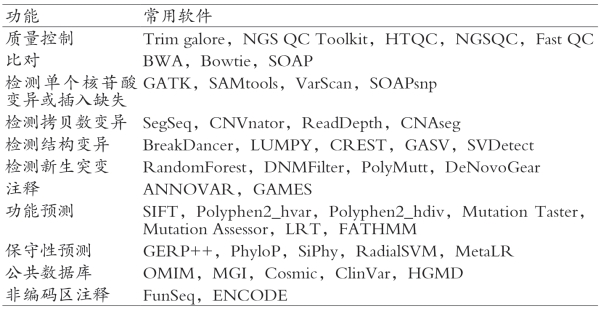
全基因组测序的数据分析流程包括质量控制(Quality control)、比对(Mapping)、突变检测(Call variant)、突变注释(Annotation)四个方面。常用的数据分析软件见表9-3,目前广泛使用的分析流程为“BWA+GATK+ANNOVAR”。
(1)质量控制:对测序产生的原始数据(Raw data)进行去接头、过滤低质量处理,得到Clean data的过程称为质量控制。质量控制能除去大约5%~15%部分测序效果较差的序列,提高后续分析的准确性。
表9-3 全基因组数据分析常用软件
(2)比对到参考基因组:将质量控制后的Clean data比对到参考基因组上,得到每条序列的比对位置、比对质量值等信息。目前最主流的比对软件为BWA(Burrows-Wheeler Aligner),它能将短序列准确快速地比对到参考基因组上。2013,BWA发布的新算法BWA MEM,可以比对70bp~1Mb的序列,结果更准确,运行速度也更快。
(3)突变检测:比对好的序列进行去重(Remove duplication)并进行突变的检测。目前主流检测软件为Genome Analysis Toolkit(GATK),通过两次校正过程以提高突变检测的准确率,准确度非常高,但是速度比较慢。2014年3月,Broad发布了最新版GATK(version3.1),在突变检测速度上比原来快3~5倍,使全基因组的分析时间从3天缩短到1天。
(4)注释突变及预测致病基因:每一个全基因组的样品,平均可以检测到大约三百万个突变。需要通过诸如ANNOVAR等软件对其进行注释,筛选致病的候选突变并用于后续功能验证,一方面,利用已知突变数据库,去除在数据库中出现频率较高的突变,将剩下的突变注释到基因组上的各个基因区间,获得突变对蛋白质编码的改变情况;另一方面,通过多个疾病数据库将部分已知突变与疾病表型联系起来,并利用预测软件对这些突变进行有害性和保守性预测,最终鉴定导致疾病发生的相关基因及突变。
(五)自动测序
DNA测序的自动化是在桑格的双脱氧链末端终止法的基础上由美国应用生物系统公司进一步发展起来的激光测序法,其基本原理与末端终止法一样,都是通过ddNTP竞争性地终止新合成的DNA链。但自动测序克服了放射性核素的污染,操作步骤繁琐、效率低和速度慢等缺点。随着计算机软件技术、仪器制造和分子生物学研究的迅速发展,DNA自动化测序技术取得了突破性进展,以其简单、安全、精确和快速等优点,已成为今天DNA序列分析的主流。
1.DNA自动测序与手工测序的不同点 ①标记物不同:手工测序采用放射性核素标记,而自动测序采用4种荧光染料分别标记ddNTP或标记引物。②加样方式不同:手工测序的一个样品的4个测序反应物分别在不同泳道进行,而自动测序可在一个泳道内同时电泳。③检测手段不同:手工测序采用放射自显影,从4种寡聚核苷酸的梯子形图谱中读出DNA序列,而自动测序则采用激光扫描器同步扫描,计算机进行阅读和编辑。
2.基本原理 自动化测序采用PCR测序模式,但只用一条引物,即在反应体系中除了加入正常反应所必需的4种dNTP外,还加入了一定比例的4种荧光染料基团标记的2',3'-ddNTP,DNA链合成过程中,dNTP和荧光染料基团标记的2',3'-ddNTP处于一种竞争状态,结果DNA合成反应的产物是一系列长度不等的具有荧光信号的多核苷酸片段。由于这4种荧光染料可激发不同颜色的荧光,4种链终止反应可在同一试管中进行并在同一泳道中判读4种碱基,不仅降低了测序泳道间迁移率差异对精确性的影响而且增加了一个电泳样品的数目,能够测定多个样品。最后借助计算机自动处理数据最终得到DNA碱基的排列顺序。常用的DNA测序仪有ABI Prism DNA Sequencer系列的377、310、3700和3730XL等型号。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。