5.1.2 结果与分析
1.腐乳中细菌DGGE图谱及分析
本研究首先使用变性梯度凝胶电泳(PCR-DGGE)技术对样品16s rRNA V3区域细菌群落组成进行研究,如图5-1所示。
由图5-1可知,图谱中共出现6条明亮的条带,同时条带1、2、4、5和6是两个样品的共有条带,说明不同腐乳样品中存在一些共有的细菌种群,其中条带1最亮且存在于两个样品中,说明此条带所对应的细菌在腐乳中发挥着重要的作用。值得一提的是各条带亮度不同,条带2和条带5在FR01中亮度较高,条带6在FR02中亮度较高,表明在细菌种群丰富度存在差异。而条带3仅存在FR02样品中,说明不同腐乳样品中存在着不同的细菌菌群。进一步将各条带进行序列分析,结果如表5-1所示。

图5-1 腐乳细菌PCR-DGGE图谱
表5-1 腐乳细菌DGGE比对结果
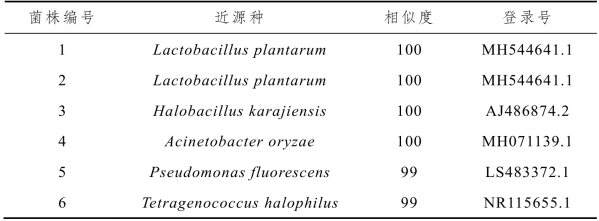
由表5-1可知,各条带序列与数据库中16s rRNA序列均具有较高的相似度。其中条带1和2为植物乳杆菌(Lactobacillus plantarum),条带3为嗜盐芽孢杆菌属细菌(Halobacillus karajiensis),条带4为不动杆菌属细菌(Acinetobacter oryzae),条带5为荧光假单胞杆菌(Pseudomonas fluorescens),条带6为嗜盐四联球菌(Tetragenococcus halophilus)。由此可知,腐乳样品中微生物构成具有多样性,而且隶属于乳杆菌属(Lactobacillus)的植物乳杆菌(Lactobacillus plantarum)为优势细菌。陈颖慧[20]利用PCR-DGGE技术对4种不同品牌腐乳中的细菌多样性进行了研究,结果发现乳酸菌属、藤黄微球菌(Micrococcus luteus)和屎肠球菌(Enterococcus Faecium)在各腐乳样品中均存在,进一步发现乳酸杆菌属(Lactobacillus)为优势细菌。陈浩[21]利用构建16s rRNA基因文库的方法对豆酱样品进行研究,结果发现嗜盐四联球菌(Tetragenococcus halophilus)为优势细菌,同时不动杆菌(Acinetobacter baylyi)也被检测到存在于样品中。王夫杰[22]在青方腐乳中分离出植物乳杆菌(Lactobacillus plantarum)和干酪乳杆菌(Lactobacillus casei)等乳酸菌。这与上述结论相一致。
本研究进一步将各条带序列与模式菌株序列进行系统发育树的构建,结果图5-2所示。
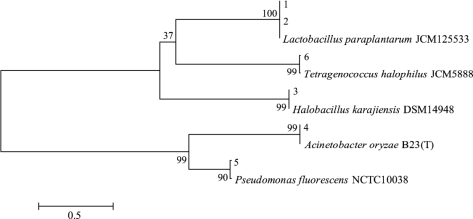
图5-2 腐乳中细菌系统发育树
由图5-2可知,系统发育树被分为2大分支,其中条带1和2与Lactobacillus plantarum聚为一类,条带6与Tetragenococcus halophilus聚为一类,条带3与Halobacillus karajiensis聚为一类。而条带4和条带5聚集在另外一支上,其中条带4和Acinetobacter oryzae聚为一类,条带5和Pseudomonas fluorescens聚为一类。由此可知。不同腐乳样品中细菌群落构成存在一定的差异性。
2.序列丰富度及多样性分析
通过Miseq高通量测序发现,两个样品共产生78 412条高质量16s rRNA序列。本研究采用两步UCLUST算法分别以100%和97%的相似度进行序列划分并建立分类操作单元(OTU),首先根据100%相似度进行序列划分得到33 254条序列,根据97%相似度进行OTU划分后得到2 317个OTU,平均每个样品1 158个OTU。当样品测序量为36 219条序列时,FR02样品具有最大的细菌物种丰富度同时细菌多样性最高,其Chao 1指数为611,Shannon指数为5.64。进一步通过稀疏曲线和香农指数曲线图对各样品产生的数据量来判定是否满足后续生物信息学分析,其结果如图5-3所示。
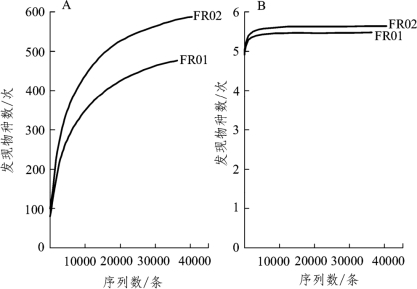
图5-3 稀疏曲线图(A)和香农指数曲线图(B)
由图5-3(A)可知,随着测序量不断的增加,各样品被发现OTU的数量也随之增加,而由图5-3(B)可知,当序列数达到10 000条时,各样品的香农多样性曲线已处于饱和状态,由此可知随着测序序列数的增加,尽管会有新的细菌种系型出现,但其多样性不再发生变化,可以反映样品中绝大多数微生物物种信息。因而本研究中每个样品产生的序列数是可以将样品中细菌微生物多样性表现出来,同时可以满足后续生物信息学分析需求。(https://www.daowen.com)
3.基于不同分类地位腐乳样品核心细菌菌群相对含量分析
纳入本研究的序列被鉴定为14个门,24个纲,53个目,84个科,145个属,其中只有5.3%的序列不能鉴定到属水平。研究发现腐乳样品中平均相对含量>1%的细菌门为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和硬壁菌门(Firmicutes),其含量分别为80.45%、10.7%和6.81%。同时在2个样品中隶属于变形菌门(Proteobacteria)的细菌相对含量分别为77.29%和83.61%,隶属于拟杆菌门(Bacteroidetes)的细菌相对含量为17.19%和4.22%,而隶属于硬壁菌门(Firmicutes)的细菌相对平均含量为3.76%和9.87%。由此可知,在门水平上各样品中的微生物存在差异,本试验进一步对各样品在属水平上进行分析,如图5-4所示。
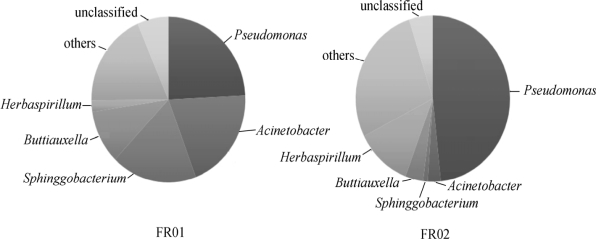
图5-4 腐乳中优势细菌属平均相对含量比较分析
由图5-4可知,腐乳样品中平均相对含量>5%的细菌属包括绿脓杆菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、鞘氨醇杆菌属(Sphingobacterium)、布丘氏菌属(Buttiauxella)和草螺菌属(Herbaspirillum),其平均相对含量分别为33%、10.56%、8.82%、6.57%和6.32%。然而各细菌属在2个样品中的相对含量均存在很大差异,其中绿脓杆菌属(Pseudomonas)的相对含量分别为22.56%和43.46%,同时不动杆菌属(Acinetobacter)的相对含量分别为18.68%和2.43%,鞘氨醇杆菌属(Sphingobacterium)的相对含量分别为16.77%和0.88%,这与PCR-DGGE结果相一致。刘亚栋[23]利用16s rDNA测序的方法对腐乳中的微生物多样性进行鉴定分析,结果发现变形菌门(Proteobacteria)、硬壁菌门(Firmicutes)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)为腐乳样品中的优势细菌门,进一步分析发现四联球菌属(Tetragenococcus)、盐单胞菌属(Halanaerobium)、Rummeliibacillus属和乳酸杆菌属(Lactobacillus)、不动杆菌属(Acinetobacter)和假单胞菌属(Pseudomonas)均为腐乳汇样品中的优势属。这与上述结论一致。
本试验进一步统计了OTU在两个样品中出现次数,如图5-5所示。
由图5-5可知,在两个腐乳样品中出现1次的OTU数量分别为1 144个和1 432个,分别占OTU总数的43.8%和54.9%,序列数分别为2183条和5 426条。同时核心OTU共有34个,占OTU总数的1.3%,包含70 802条序列。进一步分析发现有9个核心OTU的相对含量>1.5%,结果如图5-6所示。
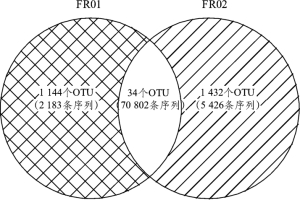
图5-5 基于OTU水平的Venn 图

图5-6 相对含量>1.5%的核心OTU热图
由图5-6可知,OTU874和OTU1711隶属于绿脓杆菌属(Pseudomonas),OTU1671和OTU894隶属于不动杆菌属(Acinetobacter),OTU577隶属于沙雷菌属(Serratia),OTU1973隶属于短波单胞菌属(Brevundimonas),OTU1907隶属于草螺菌属(Herbaspirillum),OTU635隶属于鞘氨醇杆菌属(Sphingobacterium),而OTU346未鉴定在属水平,只鉴定在Enterobacteriaceae。同时由图5-6可以发现,OTU874、OTU635和OTU1671在FR01中含量较高,其相对含量分别为16.89%、16.73%和11.32%,同时OTU874和OTU1907在FR02中含量较高,其相对含量分别为28.56%和11.85%。
4.腐乳中乳酸菌分离鉴定结果及系统发育分析
通过传统微生物培养手段结合16s rDNA测序方法对腐乳样品中的乳酸菌进行分离与鉴定,其鉴定结果和数据库中模式菌的系统发育树如图5-7所示。
图5-7 腐乳中乳酸菌系统发育树
由图5-7可知,在腐乳样品中共分离出7株乳酸菌,其中2株为戊糖片球菌(Pediococcus pentosaceus),1株为乳酸片球菌(Pediococcus acidilactici),4株为屎肠球菌(Enterococcus faecium)。由此可知,腐乳中乳酸菌种类具有多样性。
