DNA测序的一般方法
1.DNA测序的基本原理
DNA序列测定的基础是在变性聚丙烯酰胺凝胶(测序胶)上进行的高分离度的电泳过程。测序胶能在长达500 bp的单链寡核苷酸中分辨出脱氧核苷酸的差异。操作时,在相应的待测DNA区段产生一套标记的寡核苷酸单链,它们有固定的起点,但另一端按模板序列连续终止于各不相同的核苷酸。确定每个脱氧核糖核苷酸序列的关键,是在4个独立的酶学或化学反应中产生终止于所有不同的A、T、G、C位点的实核苷酸链,而这4个反应的寡核苷酸产物在测序胶的相邻泳道中都能被一一分辨出来。由于在4个泳道中再现了所有的可能寡核苷酸链,故DNA的序列能从图3-1所示的4个寡核苷酸“阶梯”中依次直接读出。
实际上,从一套测序反应中所能获得的信息量受限于测序胶的分离度。虽然最新的测序技术经常从一套测序反应中测出高达500核苷酸的信息,但获得的可靠序列信息大约为300个核苷酸。因此,如果待测DNA的区段在300核苷酸以内,那么所需的工作就是简单地将此片段克隆于合适的载体,以产生一个能方便地进行测序的重组DNA分子。
对于大片段DNA的序列测定,往往还需将其切割成能单独进行测定的小片段,这可通过随机的或有序的方式进行。下一节将讨论测定大片段DNA的策略。
目前广泛应用于DNA序列测定的方法有酶学的双脱氧法和化学裂解法,在产生寡核苷酸“阶梯”的技术上,两者截然不同。酶学双脱氧法是利用DNA聚合酶合成与模板互补的标记拷贝,化学裂解法是一套碱基专一的化学试剂作用于标记好的DNA链。下面将进一步描述DNA序列测定。
进行DNA序列测定时,4个独立的反应各产生一套放射性标记的单链寡核苷酸,它们有固定的起点,另一端终止于不同的A、T、G或C位点。每个反应的产物在高分离度的聚丙烯酰胺凝胶上电泳分级。经放射自显影,DNA序列可从凝胶上直接读出。
2.双脱氧测定法(Sanger法)
双脱氧法或酶法利用DNA聚合酶合成单链DNA模板的互补拷贝,这一方法最先由F.Sanger及其合作者于1977年提出。DNA聚合酶不能起始DNA链的合成,只能在退火于“模板”DVA的引物3′端上进行链的延伸(图3-2)。通过与模板碱基的特异性配对,脱氧核糖核苷酸(dNTP)被掺入引物的生长链。链的延伸是通过引物生长端的3羟基与被掺入脱氧核糖核苷酸的5′磷酸基的反应形成磷酸二酯键,从总体上看,链是从5′→3′方向延伸的。
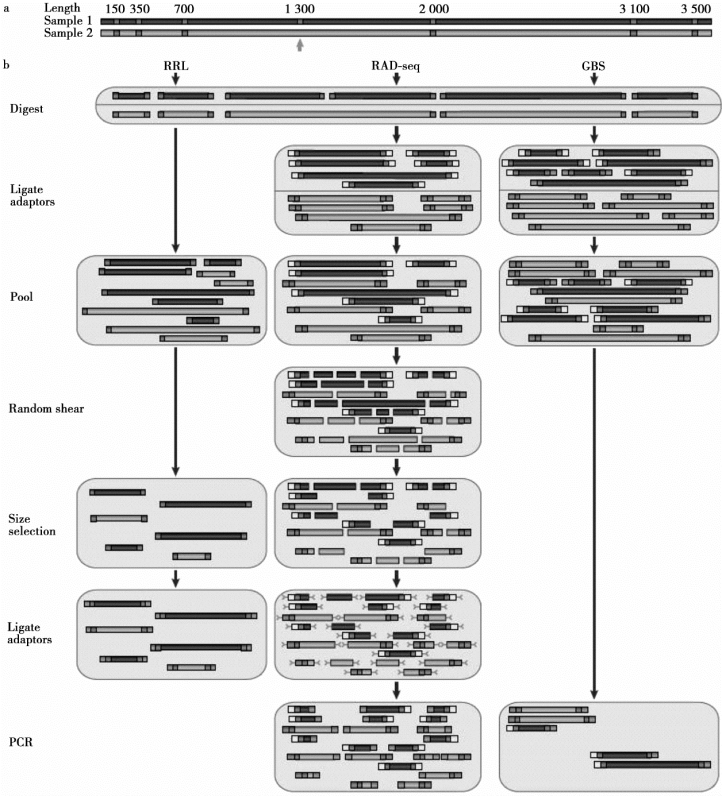
图3-1 DNA测序的一般策略
双脱氧测序法利用了DNA聚合酶能以双脱氧核糖核苷酸(ddNTP)为底物的特性。当ddNTP被掺入到延伸着的引物的3′端时,由于链上3羟基的缺如,链的延伸就终止于G、A、T或C。在4个测序反应中,每个反应只需各加入4种可能的ddNTP中的一种,就可产生如图3-2所示的4个序列阶梯。调整每个测序反应中的ddNTP与dNTP的比例,使引物的延伸在对应模板DNA上的每个可能掺入ddNTP的位置都有可能发生终止。以这种测序方式,每个延伸反应的产物都是一系列长短不一的引物延伸链,它们都具有由退火引物决定的固定的5′端以及终止于某一ddNTP的不定的3′端。
图3-2中介绍了两种双脱氧测序的工作方案。最早期的双脱氧法,本章称为Sanger法,是利用大肠杆菌DNA聚合酶Ⅰ大片段(或称Klenow片段、Klenow酶)发展起来的。“标记/终止法”则利用了一种修饰的T7DNA聚合酶,在两个独立的反应中分别进行引物的标记和双脱氧核苷酸的掺入终止。引物与模板退火后,标记反应发生在4种低浓度dNTP(其中1种是放射性标记)中,DNA的合成持续到一种或多种dNTP被耗竭为止,这样可保证掺入全部的标记的脱氧核糖核苷酸。链终止反应在4个独立的反应中进行,每个反应除了含有4种dNTP外,还各含一种ddNTP,而高浓度的dNTP保证了DNA逐次合成至生长链因ddNTP的掺入而终止。
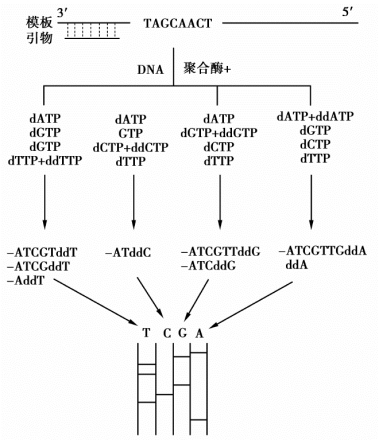
图3-2 双脱氧测序法
Sanger法测序产物的平均链长取决于ddNTP与dNTP的比例,比例高时,得到较短的产物;“标记/终止法”测序产物的平均链长可通过标记反应中dNTP浓度(高浓度能得到长的产物)或终止反应的ddNTP∶dNTP来调整。有多种商品化的用于序列测定的DNA聚合酶。热稳定的DNA聚合酶是一类最新用于测序的酶,可在高温下进行测序反应。此时DNA模板的二级结构不稳定,因而排除了它们对延伸反应的干扰。(https://www.daowen.com)
3.化学测序法(Maxam-Gilbert法)
在A.Maxam和W.Gilbert(1977)发展的DNA化学测序法中,与碱基发生专一性反应的化学试剂在一种或两种特定核苷酸位置上随机断裂已纯化的3′端或5′端标记DNA链,产生4套寡聚脱氧核糖核苷酸。在随后的测序胶放射自显影中,仅末端标记的片段显迹,故可得到如图3-3所示的4种DNA阶梯。
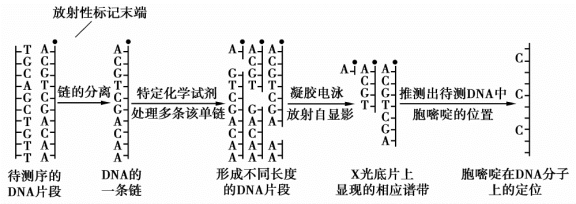
图3-3 化学测序的策略
肼、硫酸二甲酯(DMS)或甲酸可以专一性地修饰DNA分子中的碱基,这构成了化学测序法的基础,加入吡啶可催化DNA链在这些被修饰核苷酸处断裂。化学法的特异性基于第1步反应中肼、硫酸二甲酯或甲酸仅与DNA链上小部分特定碱基作用,而第2步的哌啶断裂必须定量反应。第1步反应的化学机制如下:
G反应:DMS使鸟嘌呤的7位氮原子甲基化,其后断开第8位碳原子和第9位氮原子间的化学键,哌啶置换了被修饰的鸟嘌呤与核糖的结合。
G+A反应:甲酸使嘌呤环上的氮原子质子化,削弱了腺嘌呤脱氧核糖核苷酸和鸟嘌呤脱氧核糖核苷酸中的糖苷键,然后哌啶置换了嘌呤。
T+C反应:肼断开了嘧啶环,产生的碱基片段能被哌啶置换。
C反应:在NaCl存在时,只有C才能与肼发生反应,随后被修饰的胞嘧啶被哌啶置换。
4.荧光自动测序仪
自动化测序仪使凝胶电泳、DNA条带检测和分析过程全部自动化。目前,所有的商品化DNA自动化测序仪的设计都是以Sanger法测序反应产生荧光标记或放射性标记的测序产物为基础,它们都具有数据收集的能力,并含有进一步分析处理的程序。荧光标记物通过引物或ddNTP掺入测序产物中。4种碱基产生4种颜色的荧光反应,所以用单泳道或毛细管电泳就可以分辨出相应的寡核苷酸产物。
下面结合两种型号的DNA自动测序仪介绍自动测序原理。
全自动激光荧光DNA测序系统(automated laser fluorescent DNA sequencer,ALF)是由德国海德堡(Heidelberg)欧洲分子生物学实验室(EMBL)W.Ansorge和B.Sproat提出和设计的。与同位素测序系统相比,ALF不仅在仪器硬件设计上,而且在驱控仪器的软件功能上也作了很大改进。操作中能直接分析原始数据,也可以及时处理收集过程中获取的数据。最近推出的全自动激光荧光核酸测序仪(ALF expressTM),则是利用电泳原理把荧光标记的DNA片段通过测序胶电泳分离。该仪器设计独特,能提供快速可靠的核酸测序、片段分析、HLA序列定型及突变检测等。在人类基因组大规模序列测定中,该设备起到了重要的初筛作用。ALF expressTM系统采用非放射性的单一Gy5荧光素标记引物或dNTPs进行核酸测序和片段分析,沿用Sanger双脱氧核酸末端终止测序法,使用Cy5荧光标记的引物与模板进行退火。测试时,把A、C、G、T四种反应物分别加到凝胶板上的样品槽内,上样程序与手工测序相同。另外,在仪器电泳单元的下方是由激光枪和探测器排列组成的探测系统:每个样品道后面都有一个探测器,激光能透过凝胶的每一条泳道,当DNA条带迁移到探测区域并遇上激光时,DNA上的荧光标记立刻被激活,放出荧光信号;此荧光信号由泳道前的光探测器接收,并将信息传输给电脑进行分析和保存。电泳结束后,电脑将收集到的信号(原始数据)进行处理,从而获得最终序列。
早在1987年Perkin Elmer(PE)Applied Biosystems公司就推出了DNA自动测序仪,其专利是分别采用4种荧光染料进行标记且在同一条泳道测序,具有极大的优越性。3′7型全自动DNA测序仪是PE公司近年推出的新型测序仪,它采用专利的4种荧光染料标记,并采用激光检测,具有测序精度高、每个样品判读序列长(700 bp)、一次电泳可测定样品数量多(64个)、不需要同位素测序、方法灵活多样等特点,在人类基因组测序和cDNA文库测序研究中应用极其广泛。此外,该仪器在各种应用软件的辅助下还可以进行DNA片段大小分析和定量分析,应用于基因突变分析SSCP、DNA指纹图谱分析、基因连锁图谱表达水平的研究,有着极其广泛的应用前景。其原理是采用4种荧光染料标记终止物ddNTP或引物,经Sanger测序反应后,产物3′端(标记终止物ddNTP法)或5′端(标记引物法)带有不同荧光标记,一个样品的4个测序可以在一个泳道内电泳,从而降低测序泳道间迁移率差异对精确性的影响。由于增加了一个电泳样品的数目,可一次测定64个或更多样品。经电泳后各个荧光谱带分开,同时激光检测器同步扫描,激发出的荧光经光栅分光后打到CCD摄像机上同步成像。也就是代表不同碱基信息的不同颜色荧光经光栅分光,经CCD成像,因而一次扫描可检测出多种荧光,传入电脑。与3′3型DNA测序仪相比,其测序速度大大提高,可高达200 bp/h。最终经软件分析后输出结果。
自动化测序仪的发明促进了人类基因组的大规模测序行动。自动化测序效率高,而且测序的质量也比手工操作好。由于DNA多聚酶和荧光底物不断更新,在很长一段时间内,荧光自动化测序都会处于主导地位。
