基因组测序策略
1.逐步克隆:从遗传图谱、物理图谱到基因组图谱
图3-5 限制性酶切—亚克隆法测序过程
基因组测序涉及DNA的大规模测序,它是一项如同“阿波罗登月计划”一样的庞大工程,是人类科学技术的又一次巨大进步。根据现有的技术水平,人类还无法对基因组这种复杂DNA大分子直接进行测序,而只能采取“分而治之”的测序策略,即将基因组DNA分割成一定大小的片段,然后分别对这些片段进行测序。如此便产生了这样一个问题:如何将这些片段准确地拼接起来?目前的测序方法(上节)每次反应只能测定约500 bp的DNA片段,而一般一条染色体的长度对于400~500 bp如同天文数字。所以,要对诸如人类基因组这样的基因组进行测序,则必须在两个方面取得突破:一是将基因组DNA大分子进行分割并构建适合测序的DNA片段库,而且库中的片段要覆盖整条序列;二是在整条线性序列上建立一定数量的“路标”,使切割下来的DNA片段能准确拼装回去。遗传图谱和物理图谱便是这样的“路标”图。人类遗传和物理图谱于1998年建成,并使人类基因组测序成为可能。
基因组上的DNA相当稳定,因此可以构建含有这些DNA片段的新生物体。克隆技术是把基因组上的片段插入不同生物载体,并转染到一些生物体中使其生存和稳定复制,由此可以分析由小片段DNA组成的基因组拷贝(克隆群)。目前选用插入的载体包括酵母、细菌、黏粒、噬菌体等。
遗传图谱又称连锁图谱或遗传连锁图谱,是指基因组内基因和专一的多态性DNA标记相对位置的图谱,其研究经历了从经典的基因连锁图谱到现代的DNA标记连锁图谱的过程。
构建遗传图谱的基本原理是真核生物遗传过程中会发生减数分裂,此过程中染色体要进行重组和交换,这种重组和交换的概率会随着染色体上任意两点间相对距离的远近而发生相应的变化。根据概率大小,人们就可以推断出同一条染色体上两点间的相对距离和位置关系。因此,我们得到的这张图谱就只能显示标记之间的相对距离。我们称这一距离(概率)为遗传距离,据此构建的图谱称为遗传图谱。遗传图谱的“路标”(遗传标记)已经历了几次从“粗”到“细”的大的演变,或者说,从第1代标记向第2代、第3代标记的过渡。经典的遗传标记(第1代标记)最初主要是利用蛋白质或免疫学等的标记,20世纪70年代中后期建立起来的限制性片段长度多态性(RFLP)方法成为第1代DNA标记,这类标记在整个基因组中确定的位点数目可达105以上。第2代标记为可变数量串联重复序列(variable number tandem repeat,VNTR),包括微、小卫星或短串联重复(short tandem repeat,STR或short sequent length polymorphysm,SSLP)标记等。第3代标记是称作SNP(single nuleotide polymorphysm)的遗传标记系统,即单核苷酸多态性标记。
遗传图谱上的DNA标记种类繁多,随着人类基因组等计划的进行,新的标记不断被发现,而且标记在图谱上的密度也越来越高。
遗传图谱的构建是人类基因组研究的必要步骤,它对弄清基因的功能、定位及分离克隆新基因、排列DNA片段、研究染色体上基因的排列顺序等起到了不可估量的作用。遗传图谱在人类基因组研究中发挥了巨大的作用,以致同样的策略也被复制应用于其他生物研究。
物理图谱是描述位于染色体上的基因和生物学界标独特并有确定位置及实际距离的染色体结构。任何图谱都是由一系列路标及客观物按其固有的顺序和可能的距离构建出来的。客观物的顺序不应随构图方法的改变而改变,但它们之间的距离则可能不一致。物理图谱可以理解为用物理学方法而不是遗传学方法定位的由客观物组成的任何图谱,而通常物理图谱是指高分辨率的物理图谱,即基因组长片段限制性酶切图谱和重叠克隆图谱等,但整合物理图谱还应包括只能粗略分辨路标位置但不能准确排位的染色体图谱和遗传连锁图谱。
人类基因组测序的开展还得益于以下这一系列过程的建立:脉冲场电泳(pulsed field gel electrophoresis,PFGE)技术、YAC克隆、BAC克隆和PAC克隆的出现,使切割基因组后产生的大片段DNA能准确地分离和纯化,并插入能转入DNA大片段的载体,转染酵母细胞形成YAC克隆库或转染大肠杆菌形成BAC克隆库。这些载体可载入10 Mb长度(相当于人类全基因组碱基长度的1/300)的DNA片段。全基因组的YAC克隆库及BAC克隆库保证了基因组分析的完整性和准确性。可以用杂交技术等来发现重叠克隆,以此进行克隆片段的排序。对于大片段DNA克隆进行再切割,并载入黏粒、细菌或噬菌体,即可构建相应于特定YAC或BAC克隆的亚克隆,供测序使用。
构建物理图谱最终是要统一到基于序列标签位点(sequence-tagged site,STS)的物理图谱。STS的概念最先由Olson于1989年提出,目的是建立一套统一的人类基因组生物学界标。STS本身是从人类基因组上随机选择出来的长度在200~300 bp的特异性短序列。STS路标的建立一般是从噬菌体M13上构建特定染色体克隆开始,STS概念的提出是物理构图的一次革命,由于特定STS在一套基因组结构中只出现一次,统一地把相应的克隆库中的克隆进行排序变得更准确和更科学。如果两个或两个以上的克隆包含相同的STS,则它们之间存在重叠。基于STS的物理图谱的重要性在于:①它们可用来特异地定义YAC、黏粒或噬菌体克隆;②STS可鉴定出与特定克隆存在重叠的克隆;③在计算机数据库中的各种物理图谱可以用STS通用语言统一起来。基于STS的物理图谱不但可对染色体图谱、以限制性酶切位点为路标的限制性酶切图、重叠探针杂交的YAC克隆片段重叠群图谱及其亚克隆重叠排序,以及新近发展起来的其他新方法构建的物理图谱进行整合,也可对遗传图谱、基因图谱等各类图谱进行整合,最终完成系统的、统一的基因组终极图谱。最终完成的人类基因组核苷酸序列相当于STS密度最高的基因组物理图谱。
人类基因组的各种图谱如图3.6所示。
图3-6 人类基因组的各种图谱
在上述各图谱中,最粗糙的图谱是遗传图谱,它根据相邻标记(如基因和多态片段)间的重组率来测量相互间的距离,这会造成很大的偏差;具有1~2 Mb长度的限制性酶切片段可被分离并构建物理图谱;YAC等长度在40~400 kb的插入片段排列可构建高分辨率物理图谱;碱基序列可构建最高分辨率物理图谱。
综上所述,广义上各种基于路标位点构建的物理图谱方法从低分辨率到高分辨率主要分为以下几种:(https://www.daowen.com)
①对路标进行粗略定位的染色体图谱即细胞遗传图谱,通常使用原位杂交(ISH)或荧光原位杂交(FISH)技术确定含有路标的DNA片段在染色体上的区带位置和分布。DNA片段可定位在2~10 Mb范围内。
②cDNA图谱是在细胞遗传图谱上显示cDNA或ESTs,即表达DNA(外因子)的区带位置。部分cDNA序列可作为路标。
③利用家系分离分析法可确定具有多态性的遗传标记位点在遗传连锁图谱上的位置,最新的人类基因组遗传连锁图谱已把标记间的平均距离缩小到1 cm以下,即粗略地对应于物理图谱中的1 Mb范围内。
④辐射杂种图谱是利用体细胞遗传技术构建高分辨率、长范围连续的人类基因组图谱。其基本原理为:人为地用放射线打断染色体,制备出含有特定人类染色体或片段的杂交细胞系,并利用类似于传统的减数分裂构图原理确定路标间的距离和位置。该图谱的最高分辨率可达到50 kp。
⑤脉冲场电泳的长片段限制性位点图谱,即限制性酶切位点指纹图谱,是描述以稀有酶切位点为生物学界标的顺序和距离,以及形成基因组或染色体区域上的酶切图谱。由于此方法是从DNA大片段入手,所以常常又被称为“从上到下”构图法;此外,区域性DNA大片段有利于较精细制图,如YAC克隆插入片段分析便于重叠图谱的分析,所以此方法可把DNA片段定位在100 kb~1 Mb范围内。
⑥相连组合图谱,或称重叠克隆群图谱,是由DNA片段重叠群形成的小组合,描述存在重叠的DNA片段的克隆顺序和距离。它通常通过黏粒重叠克隆把DNA片段定位在小于2 Mb的范围内,相对于长片段限制性酶切位点图谱,这种构图法也被称为“从下到上”构图法。
⑦以STS为基础的整合图,是从基因组上筛选特异序列,其最终密度至少达到平均每100 kb左右一个,最终把各种方法构建的图谱整合起来,完成准确完整的系统物理图谱。
⑧部分及全基因组测序,是分辨率最高的物理图谱,而目前要构建的高分辨率(<100 kb)物理图谱上路标序列本身也是基因组序列信息的一部分。
此外,还有一些构建物理图谱的方法,如基因组序列抽样(genomic sequence sampling,GSS)和可见图谱等。GSS是一种结合片段限制性酶切和STS的作图法,分辨率可达到1~5 kb;可见图谱则结合限制性酶切、电泳和FISH技术通过观察单个DNA大分子在限制性酶切作用下的图像来作图。
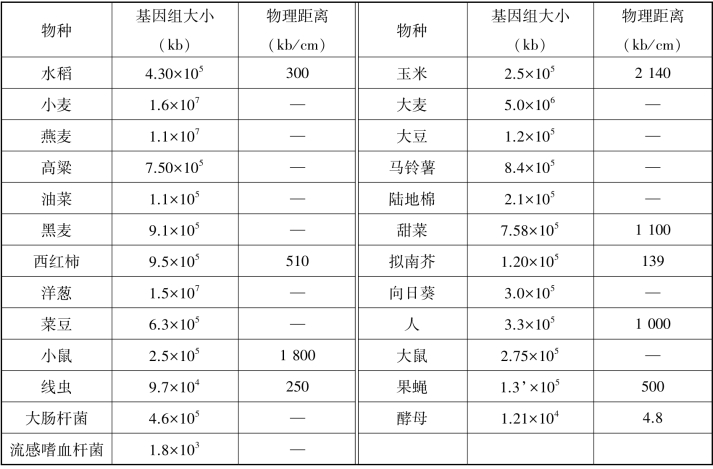
低分辨率物理图谱在人类基因组计划中本身是独立的部分,但从染色体区带—表达基因区域—遗传学距离—物理学实际距离—碱基序列这一过程来看,低分辨率染色体分带可看作粗略的物理图谱,碱基序列则是最精密的物理图谱。低分辨率图谱上的一些路标常常被用在高分辨率图谱的构建中,结合其他路标形成高密度路标分布的图谱,同时这些高密度路标可以重新在低分辨率图谱进行验证,形成高分辨率与低分辨率相结合的整合物理图谱。每种图谱都有各自的优缺点,所以即使对同一基因组进行研究,不同的实验室可能会采用不同的作图方法,但最终各种图谱的结果应能统一起来,相互补充和完善。表3-1列出了部分物种基因大小和遗传/物理距离的关系。
表3-1 部分物种基因大小和遗传/物理距离关系
可复制DNA片段作为构成物理图谱的4个基本要素之一(另3个要素是路标、单位、顺序)主要包括辐射杂种细胞(RH)、YAC、BAC、PAC等。
2.全基因组鸟枪法测序
在基因组水平上,全基因组鸟枪法和逐步克隆测定法是目前广泛应用的两个测序策略。小的单分子基因组,如细菌和小基因组(<10 Mb)可直接用鸟枪法测序。
虽然有人提出用鸟枪法直接测序人类基因组(Weber和Mayers,1997),但由于人类基因组中存在高比例的重复序列(尤其是LINE,2~7 kb)、克隆文库不可避免的间隙和基因的多态性等原因,鸟枪法的片段组装几乎是不可能的。受读序长度的限制,一个反应无法跨过LINE。鸟枪法在小基因组(1~5 Mb)测序方面已取得了非常好的效果,例如流感嗜血杆菌(H.influenzae,1.9 Mb)、支原体(M.genitalium,0.58 Mb)和甲烷球菌(M.jannaschii)基因组均用此法完成测序。逐步克隆测定法则通过建立克隆文库(YAC、BAC、PAC、Cosmid、Fosmicl、噬菌体、质粒),然后用鸟枪法进行克隆片段的测序。所以,大规模测序的两个前沿基础都是采用鸟枪法。
基因组的逐步克隆测序步骤为:DNA单链—构建BAC文库—鸟枪法克隆测序—组装;全基因组鸟枪法测序则省去中间的构建BAC文库步骤。
