基因编辑的技术原理
基因编辑技术指能够让人类对目标基因进行“编辑”,实现对特定DNA片段的敲除、加入等(图4-1)。CRISPR/Cas9技术自问世以来,就有着其他基因编辑技术无可比拟的优势,技术不断改进后,更被认为能够在活细胞中最有效、最便捷地“编辑”任何基因。
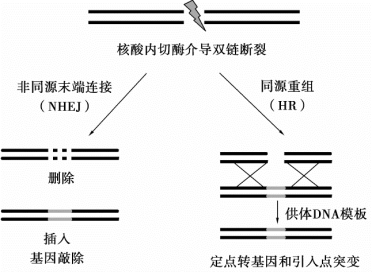
图4-1 基因编辑技术原理
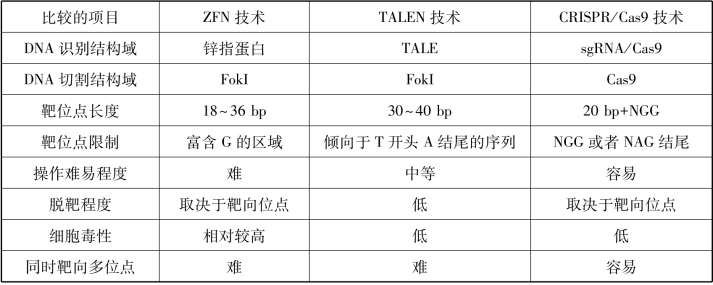
基因编辑技术主要分为3类,分别是锌指核酸酶(ZFN)技术、转录激活因子样效应物核酸酶(transcription activator-like effector nuclease,TALEN)技术以及近几年发展迅猛的CRISPR(clustered regularly interspaced short palindromic repeats,规律成簇间隔短回文重复)/Cas9技术。三种基因编辑技术的比较见表4-1。
表4-1 三种基因编辑技术比较
1.ZFN技术原理
最早出现的基因编辑技术是ZFN技术,ZFN由锌指蛋白(zinc finger protein,ZFP)和Fok I核酸内切酶两部分构成。锌指蛋白是真核生物中最丰富的一类DNA识别蛋白,Pavletich等解析了ZFP中DNA结合结构域,为设计新的DNA序列特异性结合蛋白提供了重要的基础。Sugisaki等在细菌中发现了FokI核酸内切酶,Li等发现Fok I核酸酶由DNA结合结构域和DNA切割结构域两部分组成。Chandrasegaran等用ZFP代替Fok I核酸酶的DNA结合结构域,新产生的核酸酶称为ZFN,它可以切割特异性的靶位点。ZFN的DNA识别结构域由3~4个Cys2-His2锌指蛋白串联组成,每个锌指蛋白识别一个特异的三联体碱基。多个锌指蛋白串联起来形成一个锌指蛋白组,识别一段在基因组中特异的碱基序列(9~12 bp),一个锌指蛋白组和一个FokI核酸酶相连构成一个ZFN。FokI核酸酶在二聚体状态下才有切割活性,因此需要在恰当的位置(识别位点相距5~7 bp)设计两个单体的ZFN才能切割DNA(图4-2)。ZFN技术的优点是:锌指蛋白小,编码一对ZFN只需要大约2 000 bp,这样的蛋白容易通过AAV病毒载体导入体内进行基因治疗。但是ZFN技术存在很明显的缺点,它需要一个很大的锌指蛋白库才能靶向不同的基因序列,将锌指蛋白连接在一起时,它们之间会相互干扰,影响靶向结合DNA的特异性,导致ZFN容易脱靶。因此,要想制备出高效特异的ZFN,需要大量的筛选工作,极大地阻碍了它的推广应用。设计ZFN的方法请参考OPEN和CoDA法。
2.TALEN技术原理
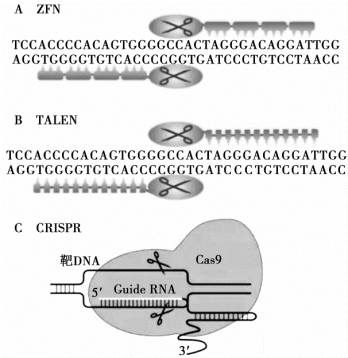
图4-2 ZFN、TALEN和CRISPR/Cas9结构的示意图
2007年,Moscou等和Boch等发现了植物黄单胞菌通过转录激活样效应因子(transcription activator-like effector,TALE)促进自身增殖的机制。黄单胞菌通过分泌系统将TALE注入植物细胞中,TALE能够靶向到启动子区域的特异DNA序列增强基因表达,这种表达反过来会促进细菌的增殖。该两团队破译了TALE识别特异DNA序列的机制,TALE依靠34个氨基酸的重复序列识别DNA序列;其中第12、13位点氨基酸为可变序列,且与碱基A、G、C和T有恒定的对应关系,即NG识别T、HD识别C、NI识别A、NN识别G,把这4种TALE模块组装起来就可以识别特异的基因组DNA序列。Cermak等把TALE模块和FokI核酸酶的切割结构域连接起来,组装成新的核酸酶叫作TALEN。TALEN的组装相对简单,活性和特异性较好。设计TALEN推荐使用Daniel Voytas实验室发明的Golden Gate组装方法。这种方法比较简单,一般的分子生物学实验室均能组装,大约需要1周的时间,用到的组装质粒可以从Addgene上获得。
3.CRISPR/Cas9技术原理
CRISPR的全称是clustered regularly interspaced short palindromic repeats(规律成簇间隔短回文重复)。CRISPR系统可分为3类(Ⅰ~Ⅲ),Ⅰ类和Ⅲ类的CRISPR系统在细菌和古生菌中均有发现,含有多个Cas蛋白;Ⅱ类CRISPR系统仅在细菌中存在,只包括一个Cas蛋白。1987年日本大阪大学的科学家在研究大肠杆菌中的碱性磷酸酶基因时,发现该基因下游存在29 bp的简单重复序列,这些重复序列被32 nt的间隔序列分开。在接下来的十多年里,类似的重复结构在越来越多的微生物和古生菌中被发现。2002年Jansen等把这种间隔重复序列命名为CRISPR,但2007年才证明CRISPR系统是细菌的一种适应性免疫系统。
Ⅱ类CRISPR系统组成最简单,除了一个Cas蛋白和crRNA(重复序列+间隔序列)外,还包括一个非编码RNA,被称为tracrRNA,它协助细菌将串联的crRNA加工成单个的crRNA,并和crRNA的重复序列互补配对后形成向导RNA,引导Cas核酸酶靶向切割外源DNA。有研究显示,酿脓链球菌中tracrRNA、crRNA和SpCas9蛋白(酿脓链球菌中的Cas称为SpCas9)3个元件在体外可以靶向切割DNA,为实现基因编辑迈出了关键的一步。之后相继有研究团队将酿脓链球菌的CRISPR/Cas9系统开发成一种可以在哺乳动物细胞中进行基因编辑的工具,成为目前应用最广泛的基因编辑技术。CRISPR/Cas9已经实现了对多个物种以及细胞系的基因编辑,如细菌、酵母、人类的癌细胞系和胚胎干细胞系、果蝇、斑马鱼、青蛙、小鼠、大鼠、兔、烟草、水稻等。
CRISPR/Cas9系统作为基因编辑工具时,crRNA和tracrRNA被融合为一条向导RNA(single-guide RNA,sgRNA)表达,所以该系统只包含sgRNA和Cas9核酸内切酶两个元件。sgRNA 5′端20 bp序列是与靶序列互补配对的序列,如果编辑某个靶位点,只需要改变这20 bp的序列就可以实现。sgRNA一般是通过人的RNA聚合酶Ⅲ启动子U6起始表达的,这个启动子起始转录的第一碱基必须是G。如果sgRNA序列第一个碱基不是G,就需要在序列前加上一个G,或者把sgRNA的第一个碱基替换成G,这样才能被U6启动子表达。这样表达的sgRNA与靶序列之间会有一个碱基不配对,但是不会影响编辑效率。CRISPR/Cas9技术的一个优点是可以在一个细胞中表达多个sgRNA,同时编辑多个靶位点,这是ZFN和TALEN无法企及的。有报道称CRISPR/Cas9系统在小鼠和斑马鱼中可以同时编辑5个基因,在大鼠细胞中可以同时编辑3个基因。
CRISPR/Cas9系统识别的位点受DNA序列的限制,不是所有的位点都可以被识别。SpCas9识别的靶序列后面必须是NGG序列,被称为PAM(protospacer-adjacentmotif)序列,因此,SpCas9识别的序列可以写成N20NGG,其中N20是与sgRNA互补配对的序列,NGG是PAM序列。在人基因组中,平均每8~12 bp就有一个GG序列。Cas蛋白不同,需要的PAM序列也不同。如果需要精确切割某个基因组位点,就可以根据基因组序列选用合适的CRISPR/Cas系统。目前被开发成基因编辑工具的CRISPR/Cas系统及其PAM序列见表4-2。
表4-2 识别不同PAM序列的CRISPR核酸酶

续表

确定好编辑的区域后,在所选区域会有很多靶位点可以选择,这时就需要选择一个最优的位点设计sgRNA。sgRNA序列与编辑的效率和特异性紧密相关。科学家通过大量的数据分析,已经找到了sgRNA序列与编辑效率之间的关系,为设计高活性的sgRNA提供了依据。除了sgRNA的活性外,还需要考虑脱靶问题。脱靶切割是基因编辑领域共同关心的一个问题,它会在基因组中引入额外的突变,影响实验结果的可靠性。sgRNA的序列与脱靶紧密相关,如果在基因组中存在与sgRNA序列相似的序列,这些位点可能也会被编辑。有研究显示,靠近PAM序列的8~12 bp对Cas9识别至关重要,这一区域的序列被称为种子序列。种子序列与sgRNA序列不匹配会严重影响Cas9核酸酶的切割;相比之下,5′端也就是远离PAM的序列具有更强的错配耐受性,即使这一区域有两三个碱基不匹配,sgRNA也有可能引导cas9核酸酶进行切割。此外,PAM序列变成NAG,Cas9也会对其进行切割,因此在检测脱靶的时候,PAM序列为NAG的相似序列,也应被考虑成潜在的脱靶序列。
脱靶问题已经成为编辑基因时必须考虑的问题。CRISPR/Cas9技术刚出现的时候,有课题组为了研究改进特异性的方法,选择了特异性差的sgRNA研究,这就造成了CRISPR/Cas9脱靶严重的印象。而后来的研究结果表明,如果sgRNA序列特异,脱靶的可能性是极其低的,甚至检测不到。基因组中只有2%的序列是编码区,即使脱靶,切割到这些区域的可能性也是极其低的,切割到这些区域而且又恰好影响到实验结果的可能性就更低了,所以大多数基因编辑领域的学者认为做基础研究时不用过分担心脱靶的问题。将来如果把CRISPR/Cas9技术用于临床治疗,脱靶问题还是需要慎重考虑的。现在有很多软件可以在线设计sgRNA,笔者推荐使用Doench等设计的网站,这个网站综合考虑了sgRNA的活性和特异性,使用人员可以提供DNA序列进行设计,也可以输入基因ID进行设计,非常方便。
