未知基因的克隆策略
和已知基因的克隆相比,未知基因的克隆难度要大很多,风险性也高。对未知基因的克隆本身就是一种探索和研究,需要研究者有更多具有创造性的研究方案和实验设计。
1.差异表达的基因克隆
生物体在不同发育阶段、不同生理状态、不同类型细胞或组织中的结构与功能的变化差异,归根结底是基因在时间与空间上的选择性表达差异,即基因差异表达。基因差异表达的研究策略主要建立在基因转录和翻译的调控过程中,即mRNA或cDNA差异和蛋白质差异两个水平,研究得较多的是mRNA或cDNA水平。通过比较不同样品的基因差异表达,分离出表达的目的基因,是一种快速分离组织特异性表达的基因的有效方法。
(1)mRNA差异显示技术
mRNA差异显示技术(mRNA differential display PCR,DDRT-PCR)是由Peng Liang等人在1992年建立的筛选基因差异表达的有效方法。它是一种将mRNA反转录技术和PCR技术相结合的RNA指纹图谱技术。
mRNA差异显示就是提取不同的总RNA,然后反转录合成cDNA。反转录时设计12种oligo(dT)12MN引物(M为A、C、G中的任意一种,N为A、C、G、T中的任意一种)。用这12种引物分别对同一总RNA样品进行cDNA合成,即进行12次不同的反转录反应,从而使反转录的cDNA具有12种类型。然后采用10个碱基组成的随机引物,对每一类cDNA进行反转录引物PCR扩增。这样通过比对不同样品的mRNA,用测序胶电泳分离PCR产物,经放射自显影即可找到被扩增的差异表达的基因。通过回收差异表达的特异条带,再次进行扩增、测序,就可以获得相关基因的序列。
mRNA差异显示技术具有简便、快速、灵敏度高和所需起始材料少的特点,此外,它还具有可以同时对多个材料或不同处理材料进行比较等优点。但mRNA差异显示具有较高频率的假阳性、重复性低、差异片段太小(多是100 bp以下),且差异大多是poly(A)尾端的非翻译区等,因此,mRNA差异显示技术筛选出真正有意义的cDNA片段并不多,虽然此技术经过了一些改进,但其缺点仍限制着此方法的充分应用。
(2)cDNA代表性差示分析
1993年Lisitsyn等建立了代表性差示分析(representational difference analysis,RDA)方法,它可以筛选出两个基因组之间的差异基因或基因片段。受Lisitsyn的启发,1994年Hubank和Schatz建立了cDNA代表性差示分析方法,此法可以筛选mRNA的差异表达。
cDNA RDA的基本原理是mRNA合成双链cDNA后,用识别4碱基的限制性内切酶进行消化。识别4碱基的限制性内切酶理论上将产生平均大小为256 bp的片段,因此可以保证绝大多数表达的基因至少有两个酶切位点,即每个基因的cDNA经识别4碱基的限制性内切酶处理的片段都带有该酶切位点,可以进行后续的扩增、消减、富集等操作。
cDNA RDA的基本步骤是:
①将对照组和实验组的双链cDNA经酶切后,两端连接上由一个12寡聚核苷酸和一个24寡聚核苷酸组成的特定12/24连接头,然后补平末端并用相应的24寡聚引物进行PCR扩增,得到具有代表性的产物——扩增子,分别称为对照组(D)和实验组(T)。扩增子的代表性在于虽然经过酶切和扩增,但仍然代表着原来cDNA样本的几乎全部信息,而且这个产物是可以扩增的。
②将T和D再分别酶切去除原来的接头,然后将T连接上新的另一不同的12/24连接头,D不连接接头进行消减杂交。消减杂交就是将T和D按1∶100~1∶800 000的比例充分混合,在一定的反应体系中充分解链和杂交,这样就形成了3种杂交体DD、TD和TT,然后再用新的12/24连接头上的24寡聚引物进行PCR扩增特异片段。DD杂交体是由D样品相同序列形成的杂交体,两头没有接头,就没有引物的结合位点,所以不能被扩增;TD杂交体是由T和D两个样品同源序列形成的杂交体,只有T一条单链上有接头序列能和引物结合,在进行PCR反应时不能有效被扩增,产物只是线性增长,PCR产物量很低;TT杂交体是由T样品相同序列形成的杂交体,两头都有接头能和引物结合,进行PCR反应时产物呈指数增长,PCR产物量高。杂交中加入过量的D样品cDNA,保证能和T样品同源的cDNA充分杂交,这样确保了TT杂交体是不同于样品D中的特异片段。杂交进行2~3轮,即对差异产物进行2~3轮的PCR富集,这样就可以得到已经富集了数百万倍的差异片段,它们会很清晰地呈现在普通琼脂糖凝胶上。
cDNA RDA进行了PCR富集,与传统的消减富集相比更加灵敏,可以筛选出低拷贝的差异基因。在操作方法上,cDNA RDA具有更加简便易行、重复性好、不需同位素、假阳性率低,在Northern印迹上重现性好等优点。但是cDNA RDA所需起始材料较多,更多依赖于PCR技术,T与D之间若存在较多差异,或T中存在某些基因上调表达,则难达到预期目的,而且工作量比DDRT-PCR大,周期长,得到的差异片段是平均为300~600 bp的小片段,还需要进一步克隆全长cDNA,在极低拷贝差异基因的筛选上还不足,即使增加PCR富集效果也还不够理想。
(3)抑制性扣除杂交
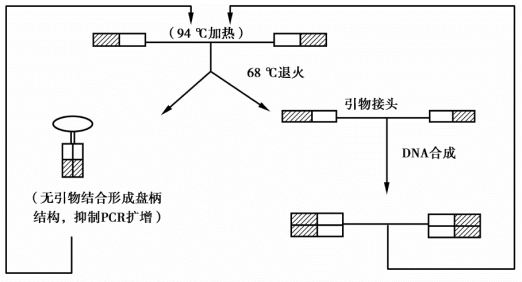
抑制性扣除杂交(suppression subtractive hybridization,SSH)由Diatchenk等于1996年提出,其技术基本原理是以抑制PCR为基础的DNA扣除杂交法,即利用非目标序列片段两端的长反向重复序列在退火时产生“锅—柄”结构,无法与引物配对,从而选择性地抑制非目标序列的扩增。同时,根据DNA分子杂交的二级动力学原理,丰度高的单链cDNA退火时产生同源杂交的速度要快于丰度低的单链cDNA,从而使具有丰度差别的cDNA相对含量基本一致。
SSH的基本过程是:分别提取T(tester)和D(driver)两种不同细胞的差异mRNA,反转录成双链cDNA,然后用RsaⅠ或HaeⅢ酶切,以产生大小适当的平头末端cDNA片段。将T的cDNA分成均等的两份,各自接上两种接头,与过量的D的cDNA变性后退火杂交,第一次杂交后有4种产物:a是T的单链cDNA,b是自身退火的T的双链cDNA,c是T和D的异源双链,d是D的cDNA。第一次杂交的目的是实现T单链cDNA均等化,即使原来有丰度差别的单链cDNA的相对含量基本一致。由于在T的cDNA中,与D的cDNA序列相似的片段大都和D形成异源双链分子c,使T的cDNA中的差异表达基因的目标cDNA得到大量富集。第一次杂交后,合并两份杂交产物,再加上新的变性D单链cDNA,再次退火杂交,此时,只有第一次杂交后经均等化和扣除的T的单链cDNA和D的单链cDNA形成各种双链分子,这次杂交进一步富集了差异表达基因的cDNA,产生了一种新的双链分子e,它的两个5′端有两个不同的接头,正是这两种不同的接头,使其在以后的PCR中被有效地扩增(图9-4)。
 (https://www.daowen.com)
(https://www.daowen.com)
图9-4 抑制PCR示意图
SSH技术可成千倍地扩增目的片段,能分离出T样品上调控表达的基因,其最大优点是假阳性率大大降低,阳性率可达94%,这是由它的两步杂交和两次PCR保证的。SSH技术进行了cDNA片段的均等化和目标片段的富集,保证了低丰度mRNA也可能被检出,使得其灵敏度高于cDNA RDA和DDRT-PCR。
但是SSH技术需要较多的起始材料,更依赖于PCR技术,不能同时进行数个材料之间或不同处理材料之间的比较。和cDNA RDA一样,需要的mRNA量高(保证有2μg以上),否则低丰度的差异表达基因的cDNA很可能会检测不到;只能对两个样品进行分析,也需要进一步获得全长的cDNA;所研究材料的差异不能太大,最好是细微差异。此外,SSH是商业公司(CLONETECH)参与研究的成果,并推出了相应的研究用途的试剂盒,但技术细节上不如cDNA RDA成熟稳定。
2.有基因图位或标记的基因克隆策略
图位克隆又称定位克隆,1986年首先由剑桥大学的Alan Coulson提出。它是一种根据目的基因在染色体上的位置进行基因克隆的方法。在不知道基因的表达产物和功能信息,又无适宜的相对表型用于表型克隆时,图位克隆是最常用的基因克隆技术,也是克隆植物基因的主要方法之一(图9-5)。
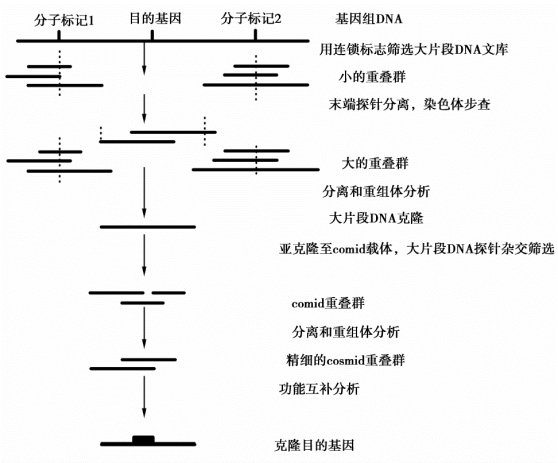
图9-5 图位基因克隆原理
图位克隆分离基因的方法:①可以根据功能基因在基因组中都有相对较稳定的基因座,再利用分离群体的遗传连锁分析将这个基因座定位到染色体的一个具体位置的基础上,通过构建高密度的分子连锁图,找到与目的基因紧密连锁的分子标记,不断缩小候选区域进而克隆到该基因;②也可以利用此物理图谱,通过染色体步移逼近目的基因或通过染色体登录的方法最终找到包含该目的基因的区域序列;③还可以使用与目的基因紧密连锁的分子标记筛选DNA文库来获得目的基因序列,最后通过遗传转化和功能互补验证,最终确定目的基因的碱基序列,并阐明其功能和疾病的生化机制。
图位克隆法分离首先要有一个根据目的基因的有无而建立起来的遗传分离群体,根据遗传分离组合关系找到与目标基因紧密连锁的分子标记,用遗传作图和物理作图将目标基因定位在染色体的特定位置;然后构建基因组文库,用分子标记筛选文库、染色体步移和亚克隆等手段获得含有目的基因的小片段克隆,再通过遗传转化和功能互补验证,最终确定目标基因的碱基序列。
图位克隆法克隆基因不仅需要构建完整的基因组文库,建立饱和的分子标记连锁图和完善的遗传转化体系,而且还要进行大量的测序工作,耗时长,工作烦琐。由于图位克隆法筛选与目标基因连锁的分子标记是克隆成功的关键,所以对基因组大、标记数目不多、重复序列较多的生物采用此法不仅投资大,而且效率低,因此图位克隆法仅应用在人类、拟南芥、水稻、番茄等图谱饱和的生物上。此外,在分析发生的变异时,可能会遇到一个性状是由不止一个基因位点控制的状况,此时利用图位克隆法来克隆此类基因变得非常困难,对其中任何一个基因位点的精细定位都要通过高代回交来创造只有一个位点保持多态性的重组近交系,这就需要花费更多的时间以及人力和物力。近年来,随着一些物种基因组测序工作的完成,各种分子标记的日趋丰富和各种数据库的完善,在此物种中用图位克隆法克隆一个基因的难度就大大降低了。
3.有转座子标记的基因克隆策略
转座(因)子是基因组中一段可移动的DNA序列,可以通过切割、重新整合等一系列过程从基因组的一个位置“跳跃”到另一个位置,这段序列称为跳跃基因或转座子。转座子可分插入序列(Is因子)、转座(Tn)和转座噬菌体,在原核微生物、真核微生物以及高等动植物都发现有转座子。
转座子标记基因克隆法是把转座子作为基因定位的标记,通过转座子在染色体上的插入和嵌合来克隆基因,这种方法在微生物的基因克隆中非常有效。转座子从一个基因位置转移到另一个位置,在转座过程中,原位置的DNA片段并未消失,发生转移的只是转座子的拷贝。基因发生转座可引起插入突变,使插入位置的基因失活并诱导产生突变型或在插入位置上出现新的编码基因,通过转座子上的标记基因(如抗药性等)就可检测出突变基因的位置和克隆出突变基因,也可以通过质粒拯救法来获得被转座子突变的基因。
利用转座子克隆植物基因的步骤主要是:先把已分离得到的转座子与选择标记构建成含转座子的质粒载体,然后把含转座子的质粒载体导入目标植物,并利用Southern杂交等技术检测转座子是否从载体质粒中转座到目标植物基因组中——这是转座子定位和分离目标基因所不可缺少的步骤,最后就是转座子插入突变的鉴定及转座子的分离和克隆。
转座子标记基因的分离和克隆的方法主要有:
①质粒拯救法:提取插入转座子植株的基因组DNA,合适的限制性内切酶消化,然后用连接酶对消化产物连接环化,再把连接产物转化大肠杆菌,利用转座子上抗性标记筛选出含有转座子的克隆,经过序列分析即可得到转座子的侧翼序列。但是质粒拯救法首先要选用合适的载体,而且不一定能得到完整的基因。
②Southern-based分离法:通过杂交得到插入转座子的纯合突变株,构建其基因组文库,以转座子的部分序列为探针从该基因文库中筛选阳性克隆,测序分析就可以得到转座子侧翼的目的基因序列,再以这一基因片段为探针,去筛选另一个正常植株的基因组文库,或者用染色体步移技术,就可以获得完整的基因。这是转座子标签法克隆基因的常用方法。
③PCR-based分离法:以插入转座子的纯合突变株的基因组DNA为PCR模板,以转座子上的已知序列设计相关引物,采用如反向PCR等的染色体步移技术或者是TAILPCR技术来获得转座子侧翼的目的基因序列,然后再在野生型中进行验证。
