从序列中寻找基因
1.基因及基因区域预测
在完成序列的拼接后,我们得到的是很长的DNA序列,甚至可能是整个基因组的序列。这些序列中包含着许多未知的基因,将未知的基因从这些序列中找出来是生物信息学的一个研究热点。
基因一词最早由丹麦生物学家W.Johannsen于1909年提出,而在这之前,遗传学创始人孟德尔用“遗传因子”表达了对基因的朦胧认识。随着遗传学、分子生物学等学科的发展,基因的概念不断得到完善。从分子生物学角度看,基因是负载特定生物遗传信息的DNA分子片段,在一定条件下能够表达特定的生物遗传信息,产生特定的生理功能。基因按其功能可分为结构基因和调控基因:结构基因可被转录形成mRNA,进而转译成多肽链;调控基因是指某些可调节控制结构基因表达的基因。在DNA链上,由蛋白质合成的起始密码开始,到终止密码子为止的连续编码序列称为开放阅读框(Open Reading Frame,ORF)。结构基因多含有插入序列,除细菌和病毒的DNA中ORF是连续的之外,包括人类在内的真核生物的大部分结构基因为断裂基因,即其编码序列在DNA分子上是不连续的,或是被插入序列隔开了(图3-7)。断裂基因被转录成前体mRNA,经过剪切过程,切除其中非编码序列(内含子),再将编码序列(外显子)连接形成成熟mRNA,并翻译成蛋白质。假基因是与功能性基因密切相关的DNA序列,但由于缺失、插入和无义突变失去阅读框而不能编码蛋白质产物。
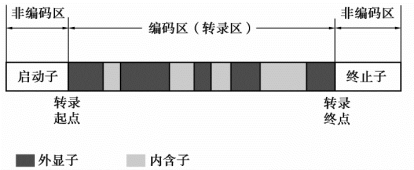
图3-7 一种典型真核蛋白质编码基因的结构示意图
基因区域预测,一般是指预测DNA序列中编码蛋白质的部分,即外显子部分。不过目前基因区域的预测已从单纯外显子预测发展到整个基因结构的预测。这些预测综合各种外显子预测的算法和人们对基因结构信号(如TATA盒等)的认识,预测出可能的完整基因。
某一算法的优劣可以通过敏感性和特异性衡量。假设待测序列中有M条序列是基因序列,而剩余的为非基因序列。我们用某一程序(算法)对待测序列进行预测,共预测出N条基因序列,而这N条序列中有N1条确实为基因。则敏感性定义为N1/M,它表示程序预测的功能;特异性定义为N1/N,它表示程序预测结果的可靠程度。敏感性和特异性往往是一对矛盾的定义。
基因区域的预测是一个活跃的研究领域,先后有一大批预测算法和相应程序被提出和应用,其中有的方法对编码序列的预测准确率高达90%以上,而且在敏感性和特异性之间取得了很好的平衡。最早的预测方法(如最长ORF法等)是通过序列核苷酸频率、密码子等特性进行预测的,随着各类数据库的建立和完善,通过相似性列线比对也可以预测可能的基因。同时,一批新方法也被提了出来,如隐马尔可夫模型(Hidden Markov Model,HMM)、动态规划法、法则系统、语言学方法、线性判别分析(Linear Discriminant Analysis,LDA)、决策树、拼接列线、傅里叶分析等。
目前基因区域预测的各种算法均基于已知基因序列。如相似性列线比较算法就是完全依赖于已知的序列,而像HMM之类的算法都需要对已知的基因结构信号进行学习或训练,由于训练所用的序列毕竟是有限的,所以对那些与学习过的基因结构不太相似的基因,这些算法的预测效果就大打折扣了。要解决以上问题,需要对基因结构进行更深入的研究,寻找隐藏在基因结构中的内在统计规律。
2.发现基因的一般过程
从序列中发现基因可以理解为基因区域预测和基因功能预测两个层次。生物信息学在这两个层次上均已形成具有自身学科特色的算法和手段,以下便简单描述通过生物信息学手段发现基因的一般过程(有关基因功能的预测将在以后的章节中进一步论述,同时本小节描述的发现过程只是生物信息学手段的一种可选策略)。
以下主要根据Gene Discovey:
·第一步:获取DNA目标序列
①如果你已有目标序列,可直接进入第二步;
②可通过PubMed查找你感兴趣的资料;也可通过GenBank或EMBL等数据库查找目标序列。
·第二步:查找ORF并将目标序列翻译成蛋白质序列
利用相应工具,如ORF Finder、Gene feature(Baylor College of Medicine)、GenLang(University of Pennsylvania)等,查找ORF并将DNA序列翻译成蛋白质序列。
·第三步:在数据库中进行序列搜索
可以利用BLAST进行ORF核苷酸序列和ORF翻译的蛋白质序列搜索。
·第四步:进行目标序列与搜索得到的相似序列的整体列线(global alignment)
虽然第三步已进行局部列线分析,但整体列线有助于进一步加深对目标序列的认识。
·第五步:查找基因家族(https://www.daowen.com)
进行多序列列线和获得列线区段的可视信息。可分别在AMAS(Oxford University)和BOXSHADE(ISREC,Switzerland)等服务器上进行。
·第六步:查找目标序列中的特定模序
①分别在Procite、BLOCK、Motif数据库进行profile、模块、模序检索;
②对蛋白质序列进行统计分析和有关预测。
·第七步:预测目标序列结构
利用PredictProtein(EMBL)、NNPREDICT(University of California)等预测目标序列的蛋白质二级结构。
·第八步:获取相关蛋白质的功能信息
为了了解目标序列的功能,收集(可利用PubMed进行)与目标序列和结构相似蛋白质的功能信息非常必要。
·第九步:把目标序列输入“提醒”服务器
如果有与目标序列相似的新序列数据输入数据库,提醒服务会向你发出通知。可选用Sequence Alerting(EMBL)、Swiss-Shop(Switzerland)等服务器。
3.解读序列
在2001年2月第二个星期里(12—18日),Science和Nature同时刊发了具有划时代意义的人类基因组研究专刊。在Science的专刊中,一篇题为“解读序列”(making sense of the sequence)的综述文章对序列,特别是人类基因组序列如何解读进行了深入分析,比较全面地展示了人类目前对序列的理解能力和技术现状。
利用基因组序列解决生物学问题已经具备了自身(学科)特色,它被冠以“功能基因组学”。自1996年酵母基因组序列被公布以来,我们已逐渐熟悉用全基因组序列来研究基因表达模式等生物学问题。尽管我们还不知道约1/3酵母基因的功能,但是我们知道所有与细胞功能有关的可能的蛋白质和RNA均由我们已知的序列编码。
根据目前基因分析的结果,哺乳动物一个基因的转录产物平均有2~3种或者更多。从现有序列数据估计,人类的基因数约为3万,这意味着人类基因组编码了约9万种或更多种蛋白质。但是,以上由现有序列数据推测的结论有很多不确定因素。重叠序列群是由单个测序反应测得的序列(通常400~800 bp)拼装而成的一条连续片段,重叠序列群的数量和长度分布是基因分析的两个重要参数。正如美国国家生物信息中心(NCBI)2000年12月12日报告所称,目前公共数据库中最大的重叠序列群为28.5 Mb,其中43个超过1 Mb,566个在250 kb和1 Mb之间,而1 628个为100~250 kb。这意味着长度大于100 kb的重叠序列群总长度约600 Mb——不足人类基因组全部序列的20%;而基因组的一半序列由22 kb或更小的重叠序列群所涵盖。由于基因的长度(一般估计为30 000碱基对)大于或等于重叠序列群,这说明一定比例的人类基因不可能只在一个重叠群中;在一个重叠群中发现一个长的基因,如肌联蛋白(Titin)基因(约250 kb,内含200多个外显子)的概率,比发现一个短的简单基因,如嗅感受蛋白基因(平均小于2 kb)的概率小得多。但要将序列缺口和重叠群扩大还要假以时日。因此,在不久的将来,基因的合成将通过组配重叠群“镶嵌物”(或称为“支架”)来完成,这意味着重叠群间的拼接又将增加序列数据的不确定性。
要想将所有的基因都落入拼装而成的无缺口的支架片段中似乎还不可能,但是组装基因的大致轮廓将变得很清楚。这就像一个被复原的古希腊花瓶,虽然花瓶的残缺部分不可能用陶土完全填补,但整个花瓶的轮廓已很清晰。在J.C.Venter等人进行基因拼装和分析的方法中,一人重要的参数是支架的大小和分布。据报道,支架的平均长度超过1 Mb,而10 Mb以上的支架占整个基因组的25%,支架间的缺口平均只有2 kb。这些为基因分析者提供了高档次的序列数据。从一给定序列片段中,通过相似性比较发现,基因的效果决定于简单的统计量和重叠群在基因组中的覆盖率。当该覆盖率达到90%以上时,那就意味着几乎所有的基因(或至少是基因片段)均可在序列数据中找到。因此,利用本周公布的数据(指Science和Nature的人类基因组研究专刊公布的数据),通过相似性搜索来发现任何一个基因几乎都是可能的。
但是必须注意的是,这样确定的基因可能还具有随意性。这是因为某一生物,例如果蝇的一条具有高度相似的受体基因序列可能来自几个不同的同源基因,而这些基因可能具有相同或完全不同的功能,甚至可能是一些没有功能的假基因。也就是说,共同的功能域或模序可能在几个基因中同时存在。使用贝尔实验室分层空时(Bell Labs Layered Space Time,BLAST)搜索工具可能是目前发现相似序列的最佳途径。NCBI网站的简明介绍有助于理解不断增多的BLAST系列工具的特性,有些小册子还介绍了BLAST近似算法的统计特色和局限。BLAST算法并不适合所有目的的近似估计,但使用者应有这样的认识:任何一种算法都有可能错过一些特殊相似性。例如,对一些相隔相似性的忽略,使间隔越大,获得相似性统计的可能性越小。新的一些方法试图利用编码区的结构因素来进行相似性比对,这突破了相似显著性方法的局限。
虽然在基因组序列基因的自动化识别方面已取得巨大进步,但根据序列构建准确的基因模型还需要大量的人力,即“手工操作”。基因的最佳模型是其全长mRNA序列。RNA序列(以cDNA形式)可以将基因组序列基因的外显子结构串联起来,而不必考虑这些片段身处何方——片段的连续性、顺序和方向并不影响串联过程。但是,假基因和高度相同的重复序列可能使这一策略失灵,这引起了对收集更多全长cDNA序列数据的争论。
大致有两条途径可以发现基因:①基于同源性的方法,包括已知mRNA序列的应用;②基因家族和特殊序列间的比较。最初的方法包括利用各种计算机手段分析外显子和其他序列信号,如酶切位点等。
在每一个基因模型中,与调控相关的序列位置和结构往往是最难完成的注释之一。在一些情况下,可以通过诸如模序(检索)来寻找和鉴定这些重要序列区段,但是我们目前对调控区段的鉴定和预测还很有限且不可靠。特定基因组间的比较是获得这些区段的一条有效途径,它建立在可以通过比较找出保守区的假设基础上。新的一些实验方法,例如列阵技术可以定位基因组水平的转录位点,同样可以有效地检测出基因组顺式调节,目前已有很多工具可以用于自动注释工作,对于这些工具的特点本文不做进一步论述。将统计学和启发式机器学习方法结合起来分析基因和基因特征是目前流行的趋势(例如隐马尔可夫模型、神经网络和贝叶斯网络)。它们发现基因最有效的方法并不是准确建模,而是常与同源性方法配合使用。影响这些算法有效性的因素包括测序误差和统计偏差,例如碱基组成。数据的噪声会极大地降低这些方法的效果,所以以上基于误差率较高的序列草图的预测结果将明显劣于基于完成序列的预测。
GENSCAN是被广泛用于基因查询和预测的软件之一,但是一些新软件,如Genie也不逊色。Genie是一种隐马尔可夫模型(HMM)系统,它可以整合不同来源的信息,如信号传感器(酶切位点、起始密码等)、内含子和外显子、mRNAEST的列线和肽序列等。其他软件工具,如GENEBUILDER、GLTMMERM、FGENES、GRAIL等,最近也都被评价过。有一个简单的办法可以比较这些软件的优劣:利用果蝇基因组数据,基因组注释评估项目(Genome Annotation Assessment Project,GASP)对真核生物基因组注释的进展和存在的问题进行很好的比较分析。另外利用拟南芥基因组也可进行相同的比较分析。
Nature和Sciece上的两篇人类基因组分析论文分别使用了各目的基因分析系统。由公共资金资助的人类基因组计划(IHGSC)使用的是一个称为“Ensembl”的系统,它使用GENSCAN进行初步预测,GENSCAN利用mRNA、EST和蛋白质模序信息进行比对;然后使用Gene Wise进行蛋白质匹配分析,Gene Wise曾被用于果蝇基因组分析。以J.C.Venter为代表的私人公司使用的是一种称为“otto”的专家注释系统,该系统力图将人的一些智能纳入程序中。
