2.1.3 Ⅱ型限制性核酸内切酶的基本特性
与Ⅰ型及Ⅲ型限制性核酸内切酶不同,Ⅱ型限制性核酸内切酶只有一种多肽,并通常以同源二聚体形式存在,因其识别序列和切割位点的特异性而在基因工程中广泛使用。
1.识别序列特异性
1)识别序列的长度
限制性核酸内切酶在双链DNA上能够识别的特殊核苷酸序列称为识别序列。为了便于书写,识别序列可以以5'→3'走向的单链DNA来表示。不同的限制性核酸内切酶各有相应的识别序列,识别序列的长度一般为4~8个碱基,最常见的为6个碱基。例如:
4个碱基识别位点:Sau3A Ⅰ GATC
5个碱基识别位点:Mae Ⅲ GTNAC(N为任意碱基)
EcoR Ⅱ CCWGG(W为A或T)
6个碱基识别位点:EcoR Ⅰ GAATTC
Hind Ⅲ AAGCTT
BamH Ⅰ GGATCC
7个碱基识别位点:BbvC Ⅰ CCTCAGC
8个碱基识别位点:Not Ⅰ GCGGCCGC
少数限制性核酸内切酶可识别两种以上的核苷酸序列,如Hind Ⅱ的识别序列为CTYRAC,其中Y为C或T,R为A或G,这样Hind Ⅱ的识别序列为四种,这种不专一性并不会影响内切酶和甲基化酶的作用位点,只是增加了DNA分子的酶识别和作用频率,为获得多种酶切片段提供了便利。
不同的限制性核酸内切酶能专一性地识别不同的核苷酸序列。但对于一个特定的限制性核酸内切酶来说,如果DNA分子中G+C的含量为50%,而且四种碱基的分布是随机的,那么根据概率统计原理,有n个碱基的识别序列的出现频率为(1/4)n。但是,实际上由于碱基排列并不完全随机,而且不同生物碱基含量不同,因此酶位点的分布及频率也不同。
2)识别序列的结构
限制性核酸内切酶识别序列的结构大多为180°旋转对称的回文结构(palindromic structure)。在一般的语言中,回文结构是指顺看、反看都一样的句子,如“step on no pets”就是一种回文体,正向阅读和反向阅读都一样。DNA的回文结构也是引申其中意义,其显著特征是在识别序列中心轴两侧的核苷酸是两两对称互补配对的,即同一条单链以中心轴对折可形成互补的双链。而且两条互补链5'到3'的序列相同(意即上面的链必须从左往右读,下面的链从右往左读),将一条链旋转180°后可与另一条链重叠。如EcoRⅠ的识别序列是:

按箭头方向,以DNA从5'到3'方向读序,两条单链顺看、反看都一样,碱基排列顺序都是GAATTC。对称轴位于第三和第四位碱基之间,其左右两侧碱基依次互补配对。(https://www.daowen.com)
2.切割位点特异性
1)切割的位置
DNA分子在限制性核酸内切酶的作用下,使多聚核苷酸链上磷酸二酯键断开的位置称为切割位点,用“↓”或“/”表示。限制性核酸内切酶在DNA上切割的位点大多数在序列内部,如EcoRⅠ(5'-G↓AATTC-3'),Ban Ⅲ(5'-AT↓CGAT-3'),Sma Ⅰ(5'-CCC ↓ GGG-3'),PstⅠ(5'-CTGCA ↓ G-3')等;也有少数则位于识别序列两侧,如Sau3A Ⅰ(5'-↓ GATC-3'),NlaⅢ(5'-CATG ↓-3'),EcoR Ⅱ(5'-↓ CCAGG-3')等。
2)产生的末端
DNA分子经限制性核酸内切酶切割后产生的DNA片段末端,通常有以下两种形式。
(1)黏性末端:切割时,DNA两条链的断裂位置是交错的,但又是对称地围绕着一个对称轴排列,产生的两个DNA末端会带有5'突出或3'突出的DNA单链,它们是相同的,也是互补的,带有这种末端的DNA分子很容易通过碱基互补配对而“黏”在一起,所以称之为黏性末端(cohesive end)或匹配黏端(matched end)。
如果在对称轴5'侧切割底物,DNA双链交错断开产生5'突出黏性末端,如EcoRⅠ:
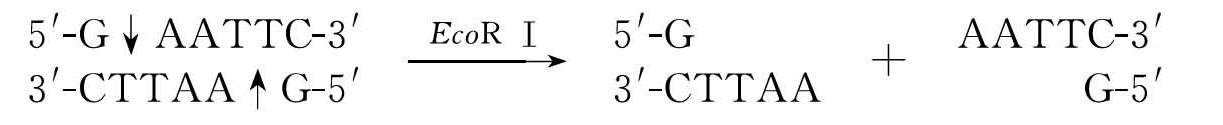
如果在对称轴3'侧切割底物,DNA双链交错断开产生3'突出黏性末端,如PstⅠ:
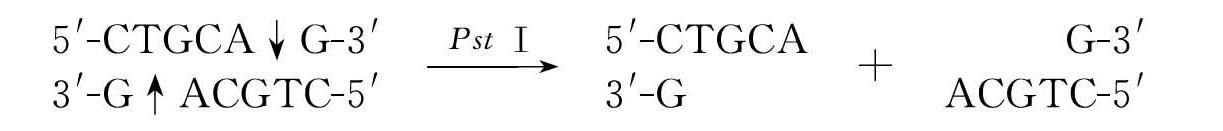
(2)平末端:切割时,若DNA两条链的断裂位置是位于回文对称轴上,产生的DNA片段是平末端(blunt end),即没有短的单链存在,如HaeⅢ和SmaⅠ。产生平末端的DNA可任意连接,但连接效率较黏性末端低。
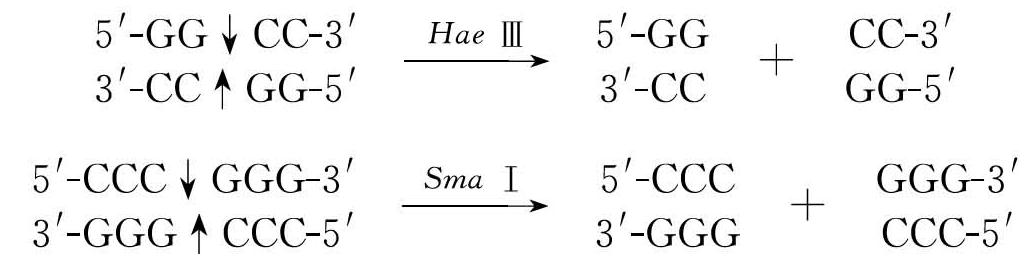
3)同裂酶
一类来源不同,但是具有相同识别序列的限制性核酸内切酶称为同裂酶(isoschizomer),也称异源同工酶。它们的切割位点可能相同或不同,具体可分为以下几种情况。
(1)识别序列相同,切割位点相同,如Sau3AⅠ(5'-↓ GATC-3')和MobⅠ(5'-↓ GATC-3'),Bam HⅠ(5'-G↓GATCC-3')和BstⅠ(5'-G ↓ GATCC-3'),Hind Ⅱ(5'-GTY ↓ RAC-3')与Hinc Ⅱ(5'-GTY↓RAC-3')(Y=C或T,R=A或G),这类酶称为同序同切酶。
(2)识别序列相同,切割位点不同,如Kpn Ⅰ(5'-GGTAC ↓ C-3')和Acc65 Ⅰ(5'-G ↓GTACC-3'),Aat Ⅱ(5'-GACGT ↓ C-3')和Zra Ⅰ(5'-GAC↓GTC-3'),Sma Ⅰ(5'-CCC↓GGG-3')和Xma Ⅰ(5'-C ↓ CCGGG-3'),这类酶称为同序异切酶。
(3)还有些识别简并序列的限制性核酸内切酶包含另一种限制性核酸内切酶的功能,如ApoⅠ(5'-R ↓ AATTY-3')(R=G或A,Y=C或T),即可识别并切割5'-G ↓ AATTC-3'、5'-A ↓ AATTC-3'、5'-G ↓ AATTT-3'、5'-A ↓ AATTT-3'四种序列,而EcoR Ⅰ(5'-G↓AATTC-3'),由此Apo Ⅰ包含EcoR Ⅰ的功能,这类酶称为同功多位酶。
4)同尾酶
有一类限制性核酸内切酶,来源不同,识别的靶序列也不同,但是它们产生相同的黏性末端,称为同尾酶(isocaudarner)。例如常用的限制性核酸内切酶BamHⅠ(5'-G↓GATCC-3')、BclⅠ(5'-T ↓ GATCA-3')、Bgl Ⅱ(5'-A ↓ GATCT-3')就是一组同尾酶,它们的识别序列和切割位点各不相同,但是切割各自识别序列的DNA后,产生相同的5'-GATC黏性末端。显而易见,这些切割产生的DNA片段通过黏性末端之间的互补作用而彼此连接起来,在基因克隆实验中很有用处。需要注意的是,由一对同尾酶分别产生的黏性末端连接后形成的新的位点,特称之为杂交位点(hybrid site)。这种杂交位点一般不能再被原来的任何一种同尾酶识别(图2-2),但也有个别情况例外,如Sau3A Ⅰ(5'-↓ GATCC-3')和Bam H Ⅰ同尾酶形成的杂交位点,对Sau3A Ⅰ仍然是敏感的,但已不再是BamH Ⅰ的靶位点。
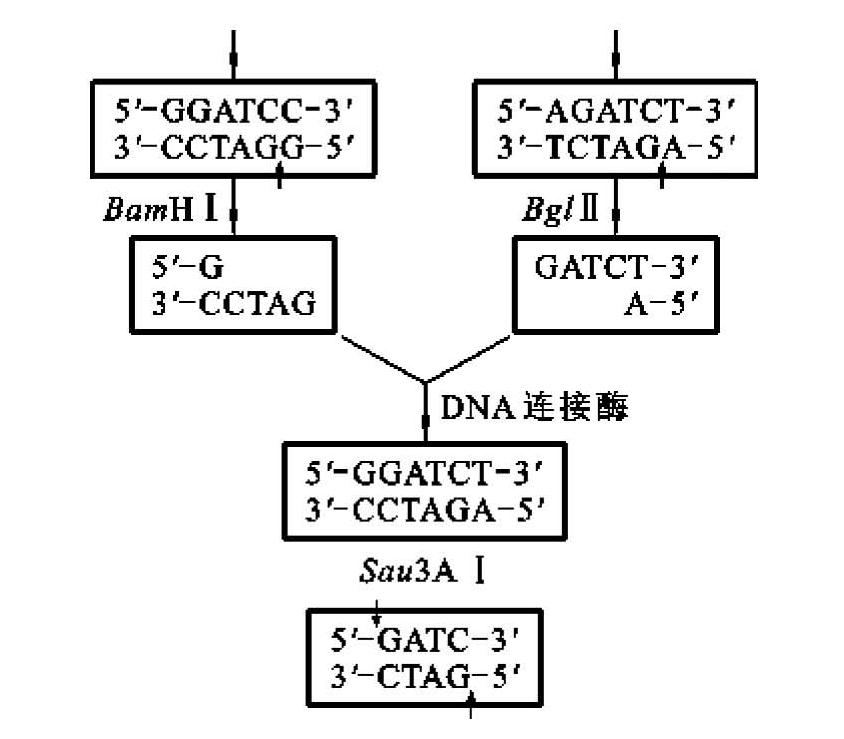
图2-2 杂交位点的形成及切割
