13.2.2 定向进化
酶的体外定向进化(directed evolution of enzyme in vitro)是改造酶蛋白质分子的一种新策略。它不需事先了解酶的定向结构和催化机制,在实验室模拟自然进化机制,通过由易错PCR、致突变菌株诱变等方法对编码酶蛋白质的基因进行随机诱变,由DNA改组(DNA shuffling)、随机引发重组和交错延伸等方法进行突变基因体外重组,设计高通量筛选方法来选出需要的突变株。它不仅可快速生产工业上有用的新酶,而且为研究蛋白质的结构与功能的关系开辟了崭新的途径,极大地拓展了蛋白质工程学的研究和应用范围。酶的体外定向进化的基本原理是,在待进化酶基因的PCR扩增反应中,利用Taq DNA聚合酶不具有3'→5'校对功能的性质,配合适当条件,如降低一种dNTP的量等,以很低的比率向目的基因中随机引入突变,构建突变库,凭借定向的选择方法,选出所需性质的优化酶(或蛋白质),从而排除其他突变体。简言之,定向进化就是随机突变加选择,前者是人为引发的,后者虽相当于环境,但只作用于突变后的分子群,起着选择某一方向的进化而排除其他方向突变的作用,整个进化过程是在人为控制下进行的。
1.定向进化的基本思路
定向进化是在不了解蛋白质的结构信息情况下,根据人为设定的目标,制备具有特定性质和功能的特殊蛋白质。在制订定向进化研究方案时需要考虑以下几个关键问题:
(1)随机诱变方法是通过引进随机的碱基替换,进而筛选理想的突变体。一般有益突变的频率很低,绝大多数突变是有害的。当突变频率太高时,几乎无法筛选到有益突变体;突变频率也不能太低,否则未发生任何突变的野生型将占据突变群体的优势,也很难筛选到理想的突变体。一般认为,理想的突变频率为每个目的基因的碱基替换在1.5~5个。
(2)组成一种蛋白质分子的氨基酸经排列组合可产生巨大的顺序空间,即可获得数量极大的排列方式,其中绝大多数排列方式没有反应功能,尤其是我们想要的功能。因此在考虑实验方案时,最好是选择一个性状最接近人们期望的酶分子作为起点。
(3)要从大量的突变体库中获得理想的个体,最重要的问题是筛选方法,筛选方法越好,成功的可能性越大。因此,应建立有效且灵敏的选择方法,确保检测出由单一氨基酸取代而引起的功能变化。
2.定向进化的常规技术
(1)易错PCR:易错PCR(error-prone PCR)是指在扩增目的基因的同时引入碱基错配,导致目的基因随机突变,改变PCR的条件,通常降低一种dNTP的量(降至5%~10%),或加入dITP来代替减少的dNTP,即会使PCR易于出错,达到随机突变的目的。然而,经一次突变的基因很难获得满意的结果,由此发展出连续易错PCR(sequential error-prone PCR)策略。即将一次PCR扩增获得的有用突变基因作为下一次PCR扩增的模板,连续反复地进行随机诱变,使每一次获得的小突变累积而产生重要的有益突变。Chen等利用此策略定向进化枯草芽孢杆菌蛋白酶E的活性获得成功,所得突变体PC3在高浓度二甲基酰胺(DMF)中,酶催化效率是野生酶的256倍。将PC3再进行两个循环的定向进化,产生的突变体13M的催化效率比PC3高3倍。用易错PCR法进行定向进化改造,关键在于突变率的控制。另外,该法较为费力、耗时,一般适用于较小的基因片段(800 bp以下)。
(2)DNA改组:DNA改组(DNA shuffling)是Stemmer于1994年建立的模仿自然进化的一种DNA体外随机突变方法。所谓DNA改组,就是将DNA拆散后重排,即将一种基因或具有结构同源型的几种基因在DNaseⅠ的作用下随机酶切成小片段,这些小片段之间有部分的碱基序列重叠,它们通过自身引导PCR(self-priming PCR)延伸,并重新组装成全长的基因,这一过程称为再组装PCR(reassembly PCR)。DNA改组的基本过程包括四个步骤(图13-12)。①目的基因的准备:根据需要选择一个基因或其片段,也可以是几个序列上具有较高同源性的基因。②DNase Ⅰ酶切:将目的基因随机切割成10~50 bp或300 bp左右的小片段。③不加引物的PCR:在Taq DNA聚合酶的作用下将DNaseⅠ切割后的DNA重叠小片段重新连接起来,在此过程中可能发生许多突变和重组。④加入引物的PCR:加入目的基因片段两端的引物,使连接好的DNA得到扩增,筛选正突变重组子。得到的正突变重组子又可以重复进行改组,使性状进一步提高。DNA改组的目的是创造将目的基因群中的突变尽可能组合的机会,导致更大的变异,最终获取最佳突变组合酶。在理论和实际上,它都优于重复寡核苷酸引导的诱变和连续易错PCR。通过DNA改组,不仅可加速积累有益突变,而且可使酶的2个或更多的已优化性状合为一体。(https://www.daowen.com)
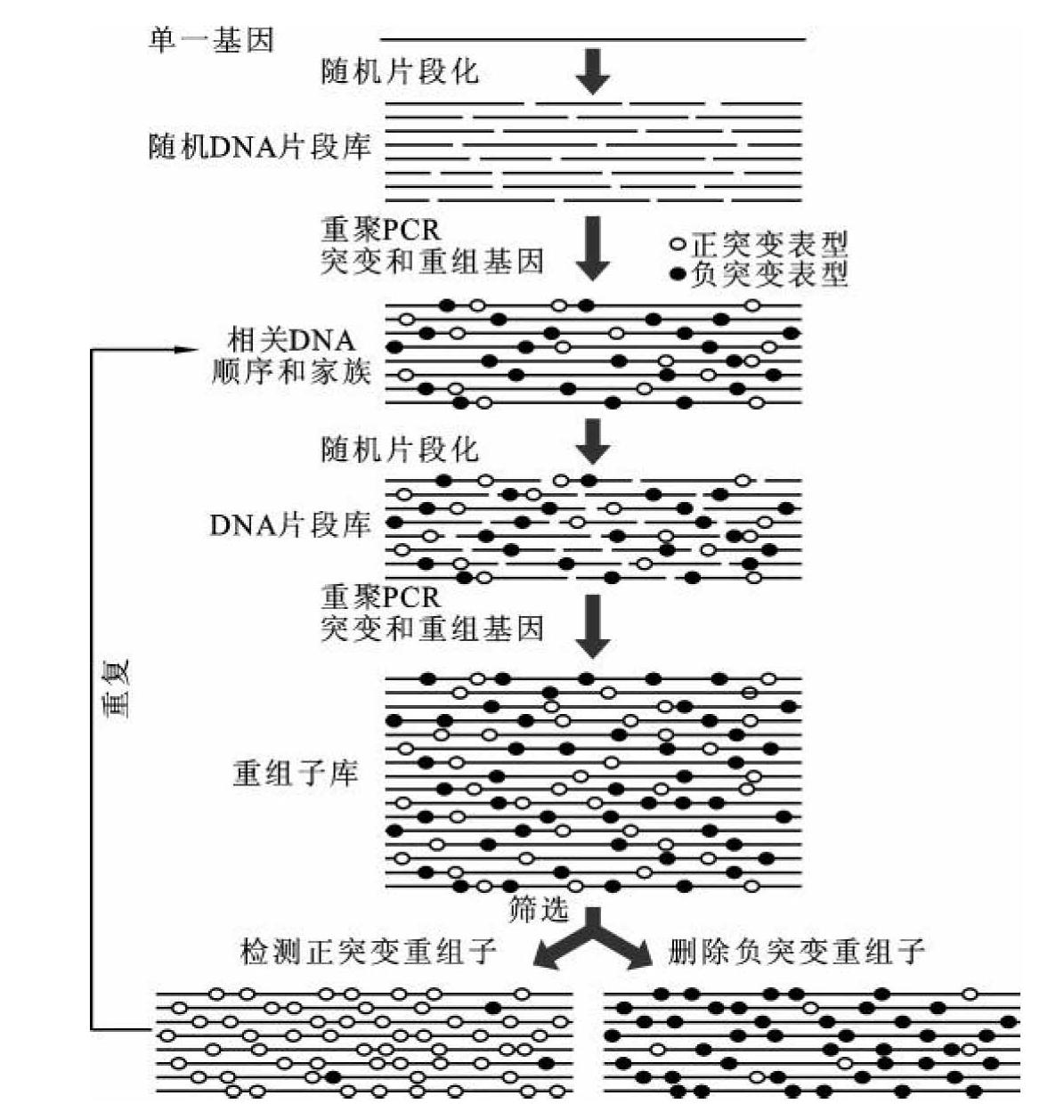
图13-12 DNA改组原图
(引自刘贤锡,2002)
(3)随机引发重组:随机引发重组(random-priming recombination,RPR)是Aronld于1998年首先报道的。其基本原理是以单链DNA为模板,配合一套随机序列引物,先产生大量互补于模板不同位点的DNA小片段,由于碱基的错误掺入和错误引发,在随后的PCR中,它们互为引物进行合成,伴随重组,再组装成完整的基因(图13-13),克隆到表达载体上,随后筛选。该法同DNA改组相比有以下优点:①RPR直接利用单链DNA或mRNA为模板;②在DNA改组中,片段重新组装前必须彻底除去DNase Ⅰ,而RPR方法省去了DNase Ⅰ切割成片段的过程,因而使基因的重组更容易;③合成的随机引物具有同样长度,无顺序倾向性,保证了点突变和交换在全长的后代基因中的随机性;④随机引发的DNA合成不受DNA模板长度的限制。
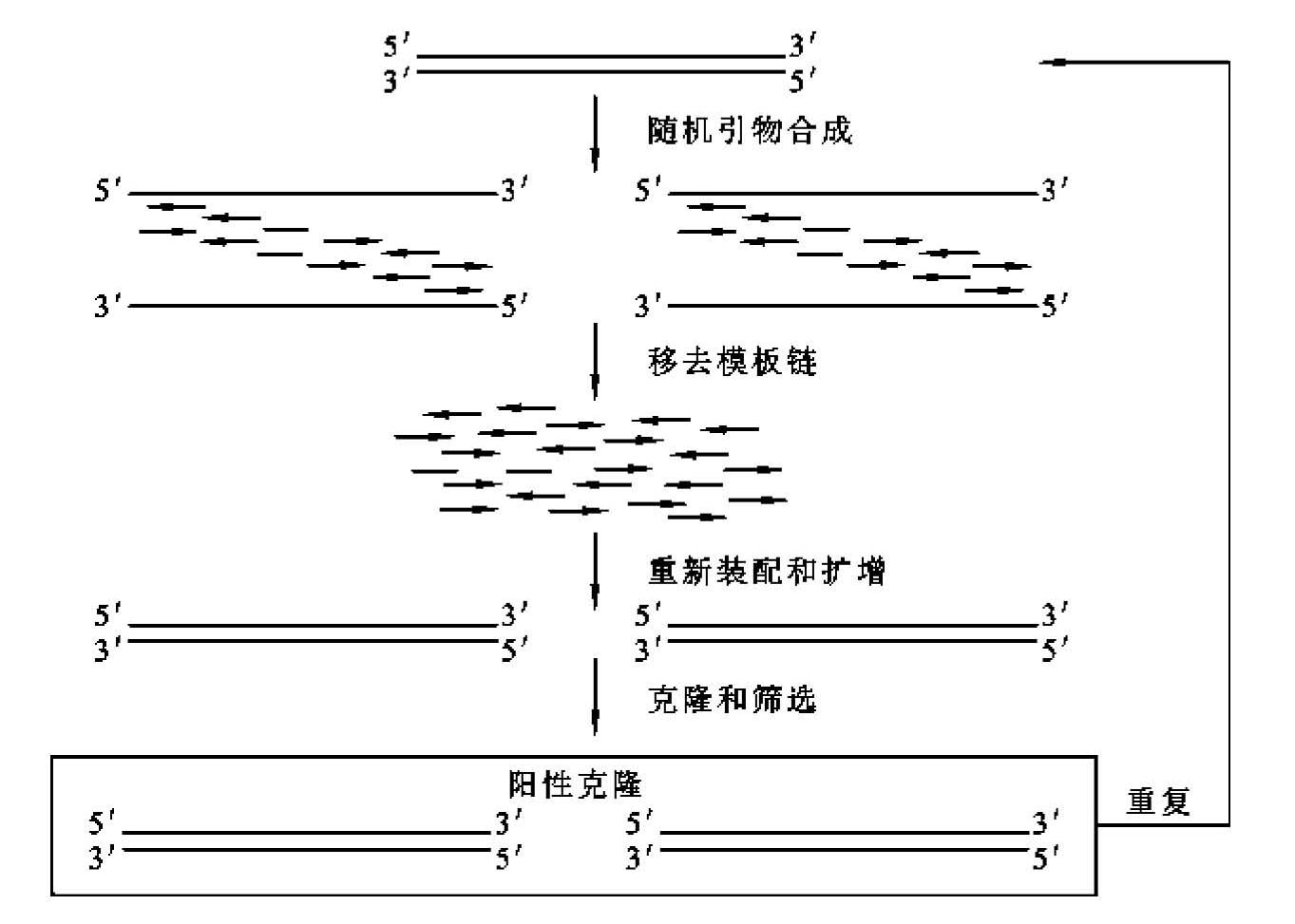
图13-13 体外随机引发重组原理
(引自刘贤锡,2002)
(4)交错延伸技术:交错延伸(stagger extension process,StEP)是Aronld等于1998年建立的一种新的体外重组方法。其原理是,在PCR中把常规的退火和延伸合并为一步,并缩短其反应时间(55℃,5s),从而只能合成出非常短的新生链,经变性的新生链再作为引物与反应体系内同时存在的不同模板退火并延伸。此过程重复进行,直到产生完整的基因长度,结果产生间隔的与不同模板序列互补的新生DNA分子(图13-14)。该技术已成功地重组了由易错PCR产生的5个热稳定的枯草芽孢杆菌蛋白酶E的突变体,得到热稳定性进一步提高的重组酶。
交错延伸法重组在单一试管中进行,不需分离亲本DNA和产生的重组DNA。它采用的是变换模板机制,这正是逆转录病毒所采用的进化过程。该法简便、有效,为酶的体外定向进化提供了又一强有力的工具。
